Détection rapide des bactéries résistantes aux antibiotiques
Une plateforme papier mise au point par des chercheurs de l'Indian Institute of Science (IISc) et du Jawaharlal Nehru Centre for Advanced Scientific Research (JNCASR) pourrait permettre de détecter rapidement la présence de bactéries pathogènes résistantes aux antibiotiques.
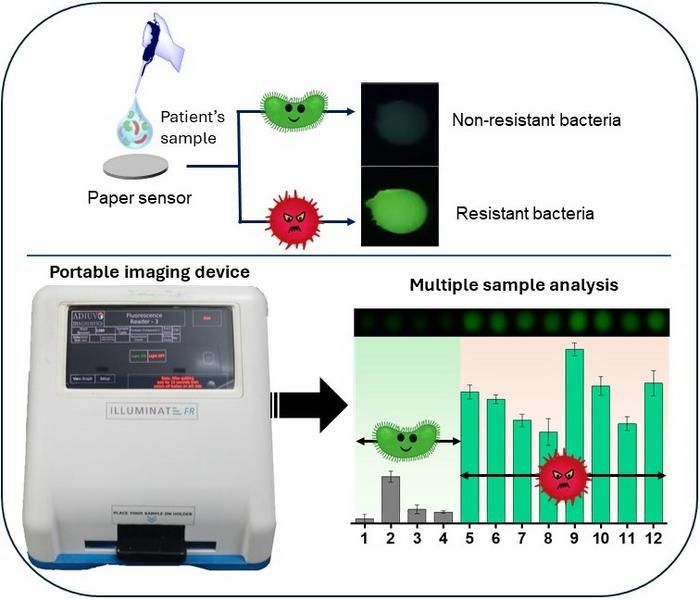
Schéma décrivant la détection/différenciation des bactéries résistantes aux antibiotiques
Arnab Dutta
L'un des plus grands défis de l'humanité est l'apparition de bactéries pathogènes résistantes aux antibiotiques. Leur apparition a été favorisée par l'utilisation abusive et excessive des antibiotiques.
Une poignée de ces bactéries, dont E. coli et Staphylococcus aureus, ont causé plus d'un million de décès, et ces chiffres devraient augmenter dans les années à venir, selon l'Organisation mondiale de la santé. Un diagnostic rapide peut améliorer l'efficacité du traitement.
"En général, le médecin diagnostique le patient et lui donne des médicaments. Le patient les prend ensuite pendant 2 ou 3 jours avant de se rendre compte que le médicament n'est pas efficace et de retourner voir le médecin. Même le fait de diagnostiquer que la bactérie est résistante aux antibiotiques à partir d'analyses de sang ou d'urine prend du temps. Nous voulions réduire ce délai de diagnostic", explique Uday Maitra, professeur au département de chimie organique de l'IISc.
Dans un article publié dans ACS Sensors, le laboratoire de Maitra et ses collaborateurs ont relevé ce défi. Ils ont mis au point un protocole de diagnostic rapide qui utilise une plate-forme de papier luminescent pour détecter la présence de bactéries résistantes aux antibiotiques.
Une bactérie peut devenir résistante aux antibiotiques de différentes manières. Dans un cas, la bactérie évolue et peut reconnaître le médicament et l'éjecter de sa cellule. Dans un autre cas, la bactérie produit une enzyme appelée β-lactamase, qui hydrolyse l'anneau β-lactame - un composant structurel clé d'antibiotiques courants comme la pénicilline et le carbapénème - rendant le médicament inefficace.
L'approche développée par l'équipe de l'IISc et du JNCASR consiste à incorporer l'acide biphényl-4-carboxylique (BCA) dans une matrice d'hydrogel supramoléculaire contenant du cholate de terbium (TbCh). Cet hydrogel émet normalement une fluorescence verte lorsqu'il est éclairé par une lumière UV.
"En laboratoire, nous avons synthétisé un substrat enzymatique en liant le BCA à l'anneau cyclique [β-lactame] qui fait partie de l'antibiotique. Lorsqu'on le mélange à un hydrogel de TbCh, il n'y a pas d'émission verte car le sensibilisateur est "masqué"", explique Arnab Dutta, doctorant au département de chimie organique de l'IISc et auteur principal de l'article. "En présence de l'enzyme β-lactamase, le gel produit une émission verte. L'enzyme β-lactamase de la bactérie est celle qui ouvre le médicament, détruit et démasque le sensibilisateur BCA. La présence de la β-lactamase est donc signalée par une émission verte". La luminescence signale la présence de bactéries résistantes aux antibiotiques, et l'intensité de la luminescence indique la charge bactérienne. Pour les bactéries non résistantes, l'intensité du vert s'est avérée extrêmement faible, ce qui permet de les distinguer plus facilement des bactéries résistantes.
L'étape suivante consistait à trouver un moyen de rendre la technologie peu coûteuse. Les instruments de diagnostic actuellement utilisés sont coûteux, ce qui fait grimper le prix des tests.
L'équipe a collaboré avec une entreprise du Tamil Nadu, Adiuvo Diagnostics, pour concevoir un appareil d'imagerie personnalisé, portable et miniature, appelé Illuminate Fluorescence Reader. L'infusion de l'hydrogel dans une feuille de papier a permis de réduire considérablement le coût de l'appareil. L'appareil est équipé de différentes LED qui émettent des rayons UV en fonction des besoins. La fluorescence verte de l'enzyme est captée par une caméra intégrée et un logiciel dédié mesure l'intensité, ce qui permet de quantifier la charge bactérienne.
L'équipe de l'IISc s'est associée au groupe de recherche de Jayanta Haldar du JNCASR pour vérifier son approche sur des échantillons d'urine. "Nous avons utilisé des échantillons de volontaires sains et ajouté des bactéries pathogènes pour simuler des infections des voies urinaires. Le résultat a été obtenu en deux heures", explique Maitra.
Dans une prochaine étape, les chercheurs prévoient de s'associer à des hôpitaux pour tester cette technologie sur des échantillons prélevés sur des patients.
Note: Cet article a été traduit à l'aide d'un système informatique sans intervention humaine. LUMITOS propose ces traductions automatiques pour présenter un plus large éventail d'actualités. Comme cet article a été traduit avec traduction automatique, il est possible qu'il contienne des erreurs de vocabulaire, de syntaxe ou de grammaire. L'article original dans Anglais peut être trouvé ici.