Jouer les dominos : comment une protéine artificielle émerge de l'assemblage de composants individuels
Un outil libre ouvre la voie à de nombreuses applications possibles dans les domaines de la biotechnologie et de la médecine
L'ingénierie ciblée de protéines artificielles aux propriétés uniques est possible grâce à une nouvelle méthode mise au point par une équipe de recherche dirigée par le professeur Dominik Niopek à l'Institut de pharmacie et de biotechnologie moléculaire (IPMB) de l'université de Heidelberg. Elle s'articule autour d'un nouveau modèle d'intelligence artificielle. Celui-ci permet de prévoir comment deux protéines doivent être assemblées au niveau moléculaire à partir de leurs parties individuelles - les sous-unités - afin de créer une nouvelle protéine fonctionnelle et ajustable. Baptisé ProDomino, cet outil à code source ouvert ouvre la voie à de nombreuses applications possibles dans les domaines de la biotechnologie et de la médecine.
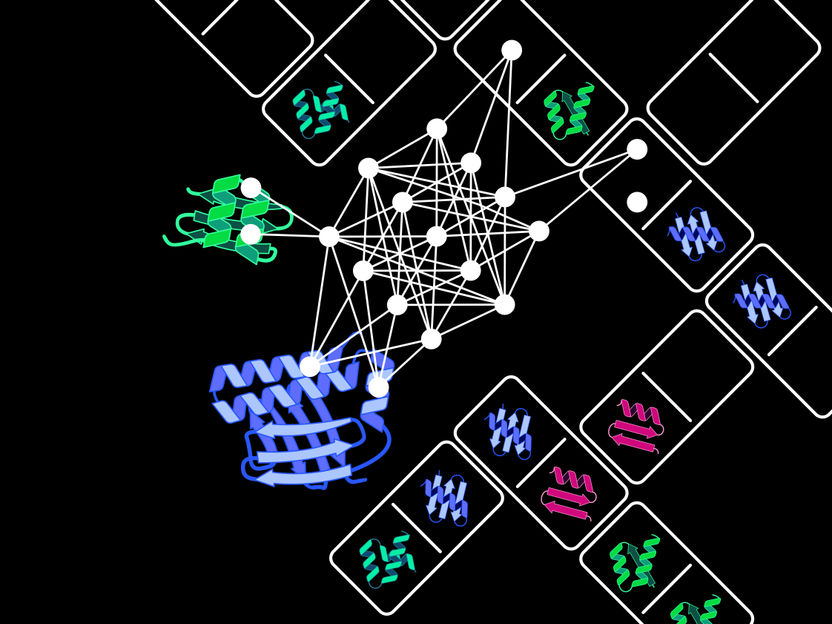
Le Protein Domain Insertion Optimizer, abrégé en ProDomino, permet de prévoir quels domaines protéiques - tels des dominos - doivent être assemblés pour créer une protéine fusionnée dotée de nouvelles propriétés.
Copyright: Jan Mathony & Benedict Wolf
Au niveau cellulaire, les protéines sont à l'origine de nombreux processus vitaux, tels que la production d'énergie ou l'augmentation du matériel génétique, et sont donc souvent appelées les "machines moléculaires" des cellules. Elles sont constituées de sous-unités interconnectées appelées domaines. "Comme les composants d'une machine technique, ces domaines ont des fonctions spécifiques. Par exemple, ils permettent aux protéines de reconnaître des signaux externes tels que la lumière ou la température, ou de catalyser des réactions chimiques avec l'aide de protéines", souligne le professeur Niopek, qui dirige le département de biologie pharmaceutique à l'IPMB. Les nouvelles protéines naissent souvent de manière naturelle lorsque des domaines existants se connectent dans de nouvelles combinaisons.
Inspirés par ces processus d'innovation évolutive, Dominik Niopek et son équipe ont développé un modèle d'IA sur la recombinaison artificielle des domaines protéiques. Pour développer et optimiser ces modèles informatiques complexes, il est essentiel de disposer de vastes ensembles de données de haute qualité. "Grâce à l'analyse automatisée d'énormes bases de données de protéines, nous avons pu produire un ensemble de données sur mesure comprenant plus de 100 000 protéines. Il reflète l'énorme variété de combinaisons de domaines naturels et a constitué la base centrale pour l'entraînement de notre modèle d'IA", explique Benedict Wolf, candidat au doctorat dans l'équipe du professeur Niopek. Le Protein Domain Insertion Optimizer, abrégé en ProDomino, peut prévoir quels domaines protéiques - tels des dominos - doivent être assemblés pour créer une protéine fusionnée dotée de nouvelles propriétés.
À l'aide de ProDomino, par exemple, des capteurs protéiques, qui réagissent à des stimuli tels que la lumière ou des substances chimiques, peuvent être combinés avec des protéines effectrices pour contrôler les processus cellulaires. Les scientifiques de Heidelberg ont ainsi réussi à coupler des domaines chimiosensibles avec les "ciseaux génétiques" CRISPR-Cas, générant ainsi des variantes CRISPR-Cas artificielles. Ces protéines hybrides peuvent être délibérément activées ou désactivées et pourraient ainsi accroître considérablement la sécurité des procédures modernes d'édition du génome, explique le Dr Jan Mathony, chef de groupe dans le département du professeur Niopek. Les chercheurs ont testé avec succès leur modèle d'IA sur différentes protéines et ont mis le logiciel à la disposition des scientifiques en tant qu'outil libre.
"Notre modèle d'IA permet de produire des protéines artificielles non seulement plus facilement, mais aussi de manière plus ciblée qu'auparavant. Cela ouvre de nouvelles perspectives pour améliorer la précision des applications basées sur les protéines dans les domaines de la biotechnologie et de la médecine, voire pour développer des approches thérapeutiques entièrement nouvelles", explique le professeur Niopek.
Note: Cet article a été traduit à l'aide d'un système informatique sans intervention humaine. LUMITOS propose ces traductions automatiques pour présenter un plus large éventail d'actualités. Comme cet article a été traduit avec traduction automatique, il est possible qu'il contienne des erreurs de vocabulaire, de syntaxe ou de grammaire. L'article original dans Anglais peut être trouvé ici.
Publication originale
Benedict Wolf, Pegi Shehu, Luca Brenker, Anna-Lisa von Bachmann, Ann-Sophie Kroell, Nicholas Southern, Stefan Holderbach, Joshua Eigenmann, Sabine Aschenbrenner, Jan Mathony, Dominik Niopek; "Rational engineering of allosteric protein switches by in silico prediction of domain insertion sites"; Nature Methods, 2025-8-4