Giocare a Dominos: come una proteina artificiale emerge dall'unione di singoli componenti
Lo strumento open-source apre molte possibilità di applicazione in biotecnologia e medicina
Annunci
L'ingegnerizzazione mirata di proteine artificiali con proprietà uniche è possibile grazie a un metodo innovativo sviluppato da un team di ricerca guidato dal Prof. Dr. Dominik Niopek presso l'Istituto di Farmacia e Biotecnologia Molecolare (IPMB) dell'Università di Heidelberg. Il metodo si basa su un nuovo modello di intelligenza artificiale. Questo modello consente di prevedere come due proteine debbano essere assemblate a livello molecolare a partire dalle singole parti - le subunità - al fine di progettare una nuova proteina funzionale e regolabile. Chiamato ProDomino, questo strumento open-source apre molte possibilità di applicazione in biotecnologia e medicina.
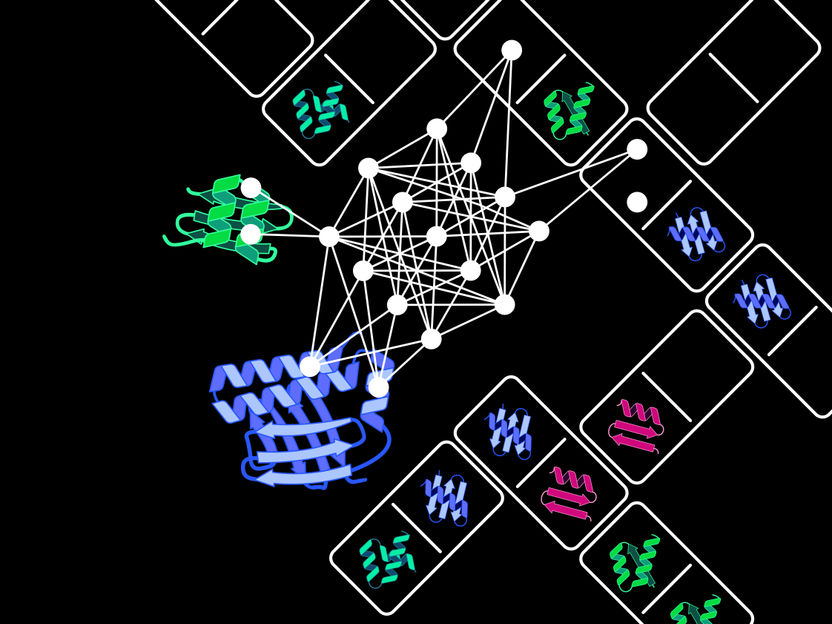
Il Protein Domain Insertion Optimizer, abbreviato in ProDomino, è in grado di prevedere quali domini proteici - come tessere del domino - devono essere incastrati tra loro per creare una proteina fusa con nuove proprietà.
Copyright: Jan Mathony & Benedict Wolf
A livello cellulare, le proteine sono alla base di numerosi processi vitali, come l'acquisizione di energia o l'incremento del materiale genetico, e per questo sono spesso chiamate "macchine molecolari" delle cellule. Sono costituite da subunità interconnesse note come domini. "Come i componenti di una macchina tecnica, questi domini hanno funzioni specifiche. Per esempio, permettono alle proteine di riconoscere segnali esterni come la luce o la temperatura, o di catalizzare reazioni chimiche con l'aiuto di proteine", sottolinea il Prof. Niopek, che dirige il dipartimento di Biologia Farmaceutica dell'IPMB. Le nuove proteine nascono spesso in modo naturale grazie alla connessione di domini esistenti in nuove combinazioni.
Ispirandosi a questi processi di innovazione evolutiva, Dominik Niopek e il suo team hanno sviluppato un modello di intelligenza artificiale sulla ricombinazione dei domini proteici. Per sviluppare e ottimizzare questi complessi modelli informatici sono essenziali set di dati estesi e di alta qualità. "Attraverso l'analisi automatizzata di enormi database di proteine, siamo riusciti a produrre un set di dati su misura con oltre 100.000 proteine. Riflette l'enorme varietà di combinazioni di domini naturali ed è stata la base centrale per l'addestramento del nostro modello di intelligenza artificiale", afferma Benedict Wolf, dottorando del team del Prof. Niopek. Il Protein Domain Insertion Optimizer, abbreviato in ProDomino, è in grado di prevedere quali domini proteici - come tessere del domino - devono essere incastrati tra loro per creare una proteina fusa con nuove proprietà.
Con l'aiuto di ProDomino, ad esempio, i sensori proteici, che reagiscono a stimoli come la luce o le sostanze chimiche, possono essere combinati con proteine effettrici per controllare i processi cellulari. Di conseguenza, gli scienziati di Heidelberg sono riusciti ad accoppiare i domini chemosensibili con le "forbici genetiche" CRISPR-Cas, generando così varianti artificiali di CRISPR-Cas. Queste proteine ibride possono essere deliberatamente attivate o disattivate e quindi potrebbero aumentare considerevolmente la sicurezza delle moderne procedure di editing del genoma, spiega il dottor Jan Mathony, leader del gruppo nel dipartimento del Prof. Niopek. I ricercatori hanno testato con successo il loro modello di intelligenza artificiale su diverse proteine e hanno messo il software a disposizione degli scienziati come strumento open-source.
"Il nostro modello di intelligenza artificiale consente di produrre proteine artificiali non solo in modo più semplice, ma anche più mirato rispetto al passato. Questo apre nuove opportunità per migliorare la precisione delle applicazioni basate sulle proteine in biotecnologia e medicina, o eventualmente per sviluppare approcci terapeutici completamente nuovi", afferma il Prof. Niopek.
Nota: questo articolo è stato tradotto utilizzando un sistema informatico senza intervento umano. LUMITOS offre queste traduzioni automatiche per presentare una gamma più ampia di notizie attuali. Poiché questo articolo è stato tradotto con traduzione automatica, è possibile che contenga errori di vocabolario, sintassi o grammatica. L'articolo originale in Inglese può essere trovato qui.
Pubblicazione originale
Benedict Wolf, Pegi Shehu, Luca Brenker, Anna-Lisa von Bachmann, Ann-Sophie Kroell, Nicholas Southern, Stefan Holderbach, Joshua Eigenmann, Sabine Aschenbrenner, Jan Mathony, Dominik Niopek; "Rational engineering of allosteric protein switches by in silico prediction of domain insertion sites"; Nature Methods, 2025-8-4