Jogando Dominó: como uma proteína artificial surge do encaixe de componentes individuais
Ferramenta de código aberto abre muitas possibilidades de aplicação em biotecnologia e medicina
Anúncios
A engenharia orientada de proteínas artificiais com propriedades únicas - é possível com a ajuda de um novo método desenvolvido por uma equipa de investigação liderada pelo Prof. Dr. Dominik Niopek no Instituto de Farmácia e Biotecnologia Molecular (IPMB) da Universidade de Heidelberg. Este método centra-se num novo modelo de IA. Este modelo permite prever a forma como duas proteínas têm de ser encaixadas a nível molecular a partir de partes individuais - subunidades - de modo a criar uma nova proteína funcional e ajustável. Denominada ProDomino, esta ferramenta de código aberto abre muitas possibilidades de aplicação em biotecnologia e medicina.
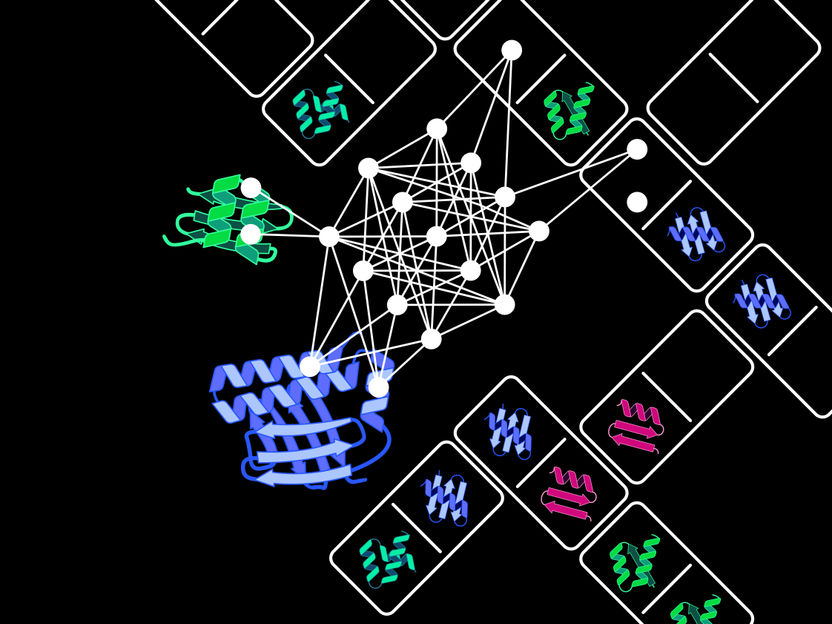
O Optimizador de Inserção de Domínios de Proteínas, abreviado como ProDomino, pode prever quais os domínios de proteínas - como peças de dominó - que têm de ser encaixados para criar uma proteína fundida com novas propriedades.
Copyright: Jan Mathony & Benedict Wolf
A nível celular, as proteínas conduzem numerosos processos vitais, como a obtenção de energia ou o aumento do material genético, pelo que são frequentemente designadas por "máquinas moleculares" das células. São constituídas por subunidades interligadas, designadas por domínios. "Tal como os componentes de uma máquina técnica, estes domínios têm funções específicas. Por exemplo, permitem que as proteínas reconheçam sinais externos, como a luz ou a temperatura, ou catalisem reacções químicas com a ajuda de proteínas", sublinha o Prof. Niopek, que dirige o departamento de Biologia Farmacêutica no IPMB. As novas proteínas surgem frequentemente de forma natural através da ligação de domínios existentes em novas combinações.
Inspirados por estes processos de inovação evolutiva, Dominik Niopek e a sua equipa desenvolveram um modelo de IA sobre a recombinação artificial de domínios proteicos. Para desenvolver e otimizar estes modelos informáticos complexos, são essenciais conjuntos de dados extensos e de elevada qualidade. "Através da análise automatizada de enormes bases de dados de proteínas, conseguimos produzir um conjunto de dados à medida com mais de 100 000 proteínas. Reflecte a enorme variedade de combinações de domínios naturais e foi a base central para o treino do nosso modelo de IA", afirma Benedict Wolf, candidato a doutoramento na equipa do Prof. Niopek. O Optimizador de Inserção de Domínios de Proteínas, abreviado como ProDomino, pode prever quais os domínios de proteínas - como peças de dominó - que têm de ser encaixados para criar uma proteína fundida com novas propriedades.
Com a ajuda do ProDomino, por exemplo, os sensores de proteínas, que reagem a estímulos como a luz ou substâncias químicas, podem ser combinados com proteínas efectoras para controlar os processos celulares. Por conseguinte, os cientistas de Heidelberg conseguiram acoplar domínios quimiossensíveis à "tesoura genética" CRISPR-Cas, gerando assim variantes artificiais de CRISPR-Cas. Estas proteínas híbridas podem ser deliberadamente activadas ou desactivadas, podendo assim aumentar consideravelmente a segurança dos procedimentos modernos de edição do genoma, explica o Dr. Jan Mathony, líder do grupo no departamento do Prof. Os investigadores testaram com êxito o seu modelo de IA em diferentes proteínas e disponibilizaram o software aos cientistas como uma ferramenta de código aberto.
"O nosso modelo de IA permite produzir proteínas artificiais não só mais facilmente, mas também de uma forma mais direcionada do que anteriormente. Isto abre novas oportunidades para melhorar a precisão das aplicações baseadas em proteínas na biotecnologia e na medicina, ou possivelmente desenvolver abordagens terapêuticas completamente novas", afirma o Prof.
Observação: Este artigo foi traduzido usando um sistema de computador sem intervenção humana. A LUMITOS oferece essas traduções automáticas para apresentar uma gama mais ampla de notícias atuais. Como este artigo foi traduzido com tradução automática, é possível que contenha erros de vocabulário, sintaxe ou gramática. O artigo original em Inglês pode ser encontrado aqui.
Publicação original
Benedict Wolf, Pegi Shehu, Luca Brenker, Anna-Lisa von Bachmann, Ann-Sophie Kroell, Nicholas Southern, Stefan Holderbach, Joshua Eigenmann, Sabine Aschenbrenner, Jan Mathony, Dominik Niopek; "Rational engineering of allosteric protein switches by in silico prediction of domain insertion sites"; Nature Methods, 2025-8-4