Jugando al dominó: cómo surge una proteína artificial a partir del encaje de componentes individuales
Una herramienta de código abierto abre numerosas posibilidades de aplicación en biotecnología y medicina
La ingeniería selectiva de proteínas artificiales con propiedades únicas es posible gracias a un novedoso método desarrollado por un equipo de investigación dirigido por el Prof. Dr. Dominik Niopek en el Instituto de Farmacia y Biotecnología Molecular (IPMB) de la Universidad de Heidelberg. Se centra en un nuevo modelo de IA. Este permite prever cómo deben encajarse dos proteínas a nivel molecular a partir de partes individuales -subunidades- para diseñar una nueva proteína funcional y ajustable. Denominada ProDomino, esta herramienta de código abierto abre numerosas posibilidades de aplicación en biotecnología y medicina.
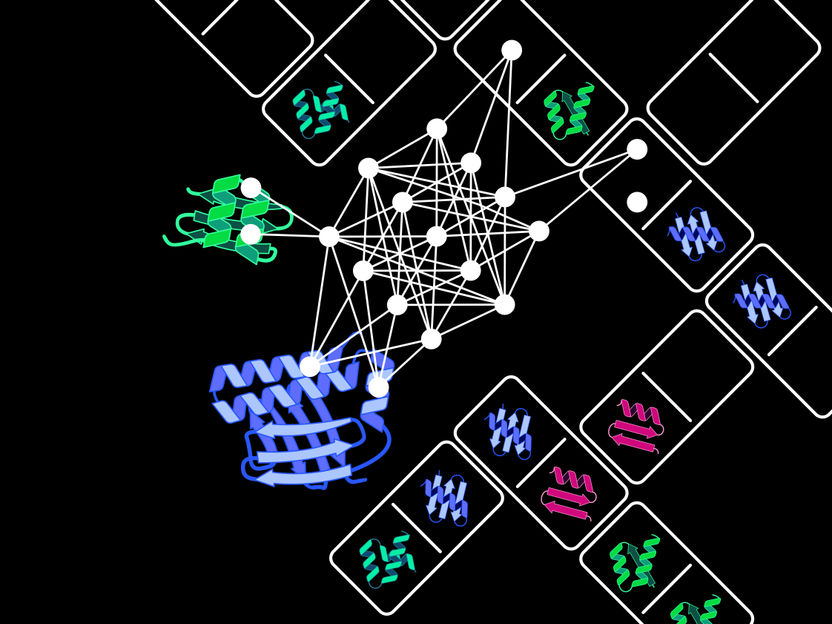
El Optimizador de Inserción de Dominios Proteicos, abreviado ProDomino, puede predecir qué dominios proteicos -como fichas de dominó- hay que encajar para diseñar una proteína fusionada con nuevas propiedades.
Copyright: Jan Mathony & Benedict Wolf
A nivel celular, las proteínas impulsan numerosos procesos vitales, como la obtención de energía o el aumento de material genético, por lo que a menudo se las denomina las "máquinas moleculares" de las células. Están formadas por subunidades interconectadas denominadas dominios. "Al igual que los componentes de una máquina técnica, estos dominios tienen funciones específicas. Por ejemplo, permiten a las proteínas reconocer señales externas como la luz o la temperatura, o catalizar reacciones químicas con la ayuda de proteínas", subraya el Prof. Niopek, que dirige el departamento de Biología Farmacéutica del IPMB. Las proteínas novedosas surgen con frecuencia de forma natural a partir de dominios existentes que se conectan en nuevas combinaciones.
Inspirándose en estos procesos de innovación evolutiva, Dominik Niopek y su equipo han desarrollado un modelo de IA sobre la recombinación artificial de dominios proteicos. Para desarrollar y optimizar estos complejos modelos informáticos son esenciales conjuntos de datos amplios y de alta calidad. "Mediante el análisis automatizado de enormes bases de datos de proteínas pudimos producir un conjunto de datos a medida con más de 100.000 proteínas. Refleja la enorme variedad de combinaciones naturales de dominios y fue la base fundamental para entrenar nuestro modelo de IA", afirma Benedict Wolf, doctorando del equipo del Prof. Niopek. El Optimizador de Inserción de Dominios Proteicos, abreviado como ProDomino, puede predecir qué dominios proteicos -como fichas de dominó- deben encajarse para diseñar una proteína fusionada con nuevas propiedades.
Con ayuda de ProDomino, por ejemplo, los sensores proteicos, que reaccionan a estímulos como la luz o sustancias químicas, pueden combinarse con proteínas efectoras para controlar procesos celulares. Así, los científicos de Heidelberg consiguieron acoplar dominios quimiosensibles con las "tijeras genéticas" CRISPR-Cas, generando así variantes artificiales de CRISPR-Cas. Estas proteínas híbridas pueden activarse o desactivarse deliberadamente, por lo que podrían aumentar considerablemente la seguridad de los modernos procedimientos de edición del genoma, explica el Dr. Jan Mathony, jefe de grupo del departamento del Prof. Niopek. Los investigadores han probado con éxito su modelo de IA en distintas proteínas y han puesto el software a disposición de los científicos como herramienta de código abierto.
"Nuestro modelo de IA permite producir proteínas artificiales no sólo con más facilidad, sino también de forma más específica que antes. Esto abre nuevas oportunidades para mejorar la precisión de las aplicaciones basadas en proteínas en biotecnología y medicina, o posiblemente para desarrollar enfoques terapéuticos completamente nuevos", afirma el profesor Niopek.
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.
Publicación original
Benedict Wolf, Pegi Shehu, Luca Brenker, Anna-Lisa von Bachmann, Ann-Sophie Kroell, Nicholas Southern, Stefan Holderbach, Joshua Eigenmann, Sabine Aschenbrenner, Jan Mathony, Dominik Niopek; "Rational engineering of allosteric protein switches by in silico prediction of domain insertion sites"; Nature Methods, 2025-8-4







































