Detección rápida de bacterias resistentes a los antibióticos
Una plataforma de papel desarrollada por investigadores del Instituto Indio de Ciencias (IISc) y el Centro Jawaharlal Nehru de Investigación Científica Avanzada (JNCASR) podría ayudar a detectar rápidamente la presencia de bacterias resistentes a los antibióticos y causantes de enfermedades.
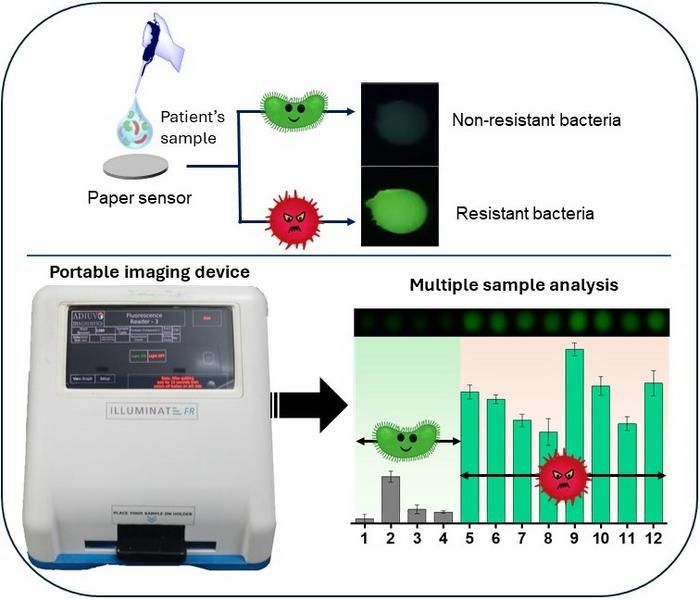
Esquema de la detección/diferenciación de bacterias resistentes a los antibióticos
Arnab Dutta
Uno de los mayores retos de la humanidad ha sido la aparición de bacterias patógenas resistentes a los antibióticos. Su aparición se ha visto favorecida por el uso indebido y excesivo de antibióticos.
Un puñado de bacterias de este tipo, como E. coli y Staphylococcus aureus, han causado más de un millón de muertes, y se prevé que esta cifra aumente en los próximos años, según la Organización Mundial de la Salud. Un diagnóstico a tiempo puede mejorar la eficacia del tratamiento.
"Generalmente, el médico diagnostica al paciente y le da medicamentos. El paciente los toma durante 2-3 días antes de darse cuenta de que el medicamento no funciona y vuelve al médico. Incluso diagnosticar que la bacteria es resistente a los antibióticos a partir de análisis de sangre u orina lleva tiempo. Queríamos reducir ese tiempo de diagnóstico", explica Uday Maitra, profesor del Departamento de Química Orgánica del IISc.
En un artículo publicado en ACS Sensors, el laboratorio de Maitra y sus colaboradores han abordado este reto. Han desarrollado un protocolo de diagnóstico rápido que utiliza una plataforma basada en papel luminiscente para detectar la presencia de bacterias resistentes a los antibióticos.
Una bacteria se vuelve resistente a los antibióticos de distintas maneras. En una, la bacteria evoluciona y puede reconocer y expulsar el medicamento fuera de su célula. En otra, la bacteria produce una enzima llamada β-lactamasa, que hidroliza el anillo β-lactámico -un componente estructural clave de antibióticos comunes como la penicilina y el carbapenem- haciendo ineficaz el medicamento.
El método desarrollado por el equipo del IISc y el JNCASR consiste en incorporar ácido bifenil-4-carboxílico (BCA) dentro de una matriz de hidrogel supramolecular que contiene colato de terbio (TbCh). Este hidrogel emite normalmente fluorescencia verde cuando se le ilumina con luz ultravioleta.
"En el laboratorio sintetizamos un sustrato enzimático uniendo BCA al anillo cíclico [β-lactámico] que forma parte del antibiótico. Al mezclarlo con el hidrogel TbCh, no se produce emisión verde, ya que el sensibilizador queda 'enmascarado'", explica Arnab Dutta, estudiante de doctorado del Departamento de Química Orgánica del IISc y autor principal del artículo. "En presencia de la enzima β-lactamasa, el gel produce una emisión verde. La enzima β-lactamasa de la bacteria es la que corta el fármaco, destruye y desenmascara el sensibilizador BCA. Por lo tanto, la presencia de β-lactamasa está señalada por la emisión verde". La luminiscencia señala la presencia de bacterias resistentes a los antibióticos, y la intensidad de la luminiscencia indica la carga bacteriana. En el caso de las bacterias no resistentes, se comprobó que la intensidad del verde era extremadamente baja, lo que facilitaba distinguirlas de las bacterias resistentes.
El siguiente paso era encontrar la forma de que la tecnología fuera barata. Los instrumentos de diagnóstico que se utilizan actualmente son caros, lo que encarece las pruebas.
El equipo colaboró con una empresa de Tamil Nadu llamada Adiuvo Diagnostics para diseñar un dispositivo de imagen personalizado, portátil y en miniatura, llamado Illuminate Fluorescence Reader. La infusión del hidrogel en una hoja de papel como soporte redujo considerablemente el coste. El instrumento está equipado con diferentes LED que emiten radiación UV según sea necesario. La fluorescencia verde de la enzima es captada por una cámara integrada y una aplicación informática específica mide la intensidad, lo que puede ayudar a cuantificar la carga bacteriana.
El equipo del IISc colaboró con el grupo de investigación de Jayanta Haldar, del JNCASR, para comprobar su método en muestras de orina. "Utilizamos muestras de voluntarios sanos y añadimos bacterias patógenas para imitar las infecciones del tracto urinario. El resultado se obtuvo en dos horas", explica Maitra.
El siguiente paso de los investigadores es colaborar con hospitales para probar esta tecnología con muestras de pacientes.
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.