Búsqueda de nuevos antibióticos sin células
Un nuevo método combina la biología sintética con la inteligencia artificial
Anuncios
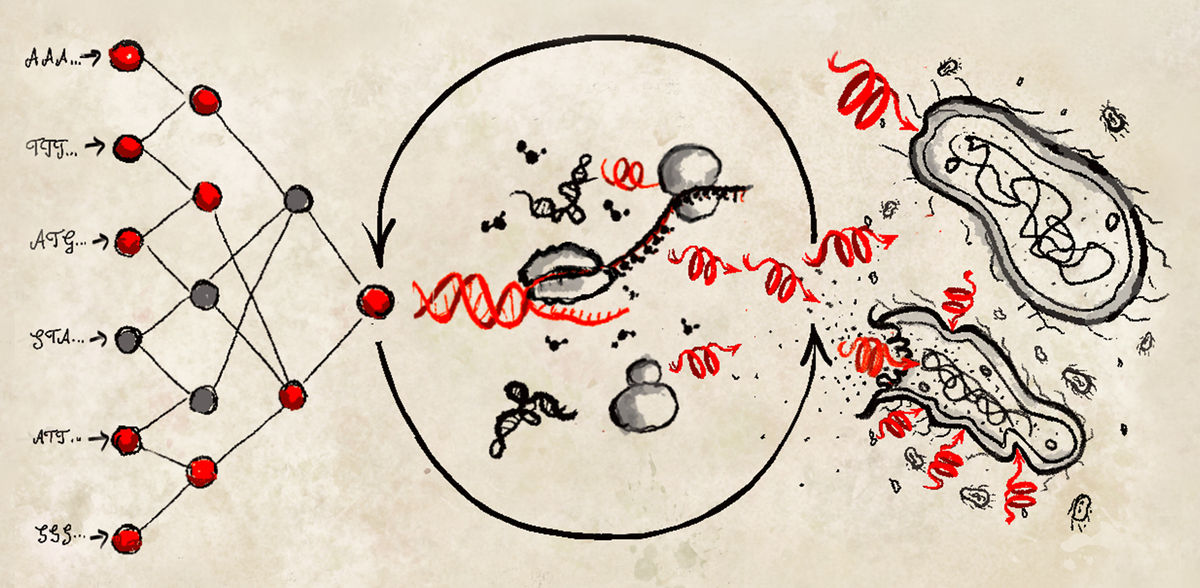
La creciente resistencia de las bacterias a los antibióticos supone un riesgo sanitario mundial cada vez mayor. Ahora, investigadores del Instituto Max Planck de Microbiología Terrestre han combinado la biología sintética y la inteligencia artificial (IA) para desarrollar un método más eficiente de búsqueda y creación de nuevos péptidos antimicrobianos eficaces contra una amplia gama de bacterias.
Los péptidos bioactivos desempeñan un papel clave en la salud y la medicina. En la actualidad se utilizan más de 80 fármacos basados en péptidos, todos ellos aislados de fuentes naturales. Sin embargo, se calcula que la resistencia a los antibióticos causa más de un millón de muertes al año en todo el mundo. Se prevé que esta cifra aumente a 10 millones en 2050, lo que crea una necesidad urgente de métodos novedosos para acelerar el desarrollo de nuevos antimicrobianos. Un potencial sin explotar se encuentra en el espacio no natural, donde se calcula que aún queda por explorar un número de entre2010 y 2030 péptidos diferentes.
En colaboración con varios laboratorios del MPI de Microbiología Terrestre, la Universidad de Marburgo, el MPI de Biofísica, el Instituto de Microbiología del Bundeswehr, el Instituto iLung y el INRAe de Francia, un equipo de científicos del Instituto Max Planck dirigido por el profesor Tobias Erb ha establecido una nueva vía para el desarrollo de péptidos bioactivos.
"En el aprendizaje profundo, una red neuronal -algoritmos inspirados en el cerebro humano- aprende a partir de grandes cantidades de datos. Este tipo de aprendizaje automático es muy prometedor para el descubrimiento de péptidos y el diseño de novo. Sin embargo, suele ir seguido de la síntesis química de péptidos para la validación experimental, que es bastante difícil y requiere mucho tiempo y limita severamente el número de péptidos que se pueden sintetizar químicamente", explica Amir Pandi, autor principal del estudio.
Para superar estas limitaciones, el equipo de investigación estableció un proceso de síntesis de proteínas sin células (CFPS) para la producción rápida y rentable de péptidos antimicrobianos (AMP) directamente a partir de plantillas de ADN. El nuevo protocolo proporciona un método rápido, de bajo coste y alto rendimiento para el cribado de AMP.
El equipo utilizó primero el aprendizaje profundo generativo para diseñar miles de AMP de novo y, a continuación, el aprendizaje profundo predictivo para reducirlos a 500 candidatos. Entre ellos, el cribado con el método sin células identificó 30 AMP funcionales, que los investigadores caracterizaron mediante simulaciones de dinámica molecular, actividad antimicrobiana y toxicidad.
En particular, seis de los AMP de novo mostraron una actividad de amplio espectro frente a patógenos multirresistentes y no desarrollaron resistencia bacteriana.
"Nos hemos beneficiado enormemente de esta combinación de biología sintética sin células, inteligencia artificial y un enfoque de alto rendimiento. Al aumentar el número de candidatos que pueden probarse experimentalmente en menos de 24 horas, aumentaron las posibilidades de encontrar AMP activos", afirma Amir Pandi. "Así pues, nuestro pipeline CFPS no sólo complementa los recientes avances en diseño computacional. También tiene el potencial de explorar la relación entre el diseño y la función de los péptidos bioactivos de forma más rápida y rentable." Tobias Erb añade: "Este nuevo método en la interfaz de la biología sintética y el aprendizaje automático será de interés para los científicos que trabajan en los campos de la biomedicina y los péptidos bioactivos."
Los próximos pasos incluyen seguir mejorando el rendimiento de la producción de péptidos, así como emplear enfoques de IA y biología sintética para diseñar nuevos AMP más estables, menos tóxicos o que añadan un modo de acción específico. Los investigadores también planean aplicar modelos generativos profundos aumentados en los que la máquina aprenda representaciones moleculares para las propiedades deseadas, lo que mejoraría la tasa de éxito en la identificación de candidatos activos.
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.