La recherche de nouveaux antibiotiques sans cellules
Une nouvelle méthode combine la biologie synthétique et l'intelligence artificielle
La résistance croissante des bactéries aux antibiotiques représente un risque de plus en plus important pour la santé mondiale. Aujourd'hui, des chercheurs de l'Institut Max Planck de microbiologie terrestre ont combiné la biologie synthétique et l'intelligence artificielle (IA) pour mettre au point une approche plus efficace de la recherche et de la création de nouveaux peptides antimicrobiens efficaces contre un large éventail de bactéries.
Les peptides bioactifs jouent un rôle clé dans la santé et la médecine. Plus de 80 médicaments à base de peptides sont actuellement utilisés, tous isolés à partir de sources naturelles. Cependant, on estime que la résistance aux antibiotiques est à l'origine de plus d'un million de décès dans le monde chaque année. Ce chiffre devrait atteindre 10 millions d'ici 2050, ce qui crée un besoin urgent de nouvelles méthodes pour accélérer le développement de nouveaux antimicrobiens. Un potentiel inexploité réside dans l'espace non naturel, où un nombre estimé de2010 à2030 peptides différents doit encore être exploré.
En collaboration avec plusieurs laboratoires du MPI pour la microbiologie terrestre, de l'université de Marbourg, du MPI pour la biophysique, de l'Institut de la Bundeswehr pour la microbiologie, de l'Institut iLung et de l'INRAe France, une équipe de scientifiques de l'Institut Max Planck dirigée par le professeur Tobias Erb a mis en place un nouveau pipeline pour le développement de peptides bioactifs.
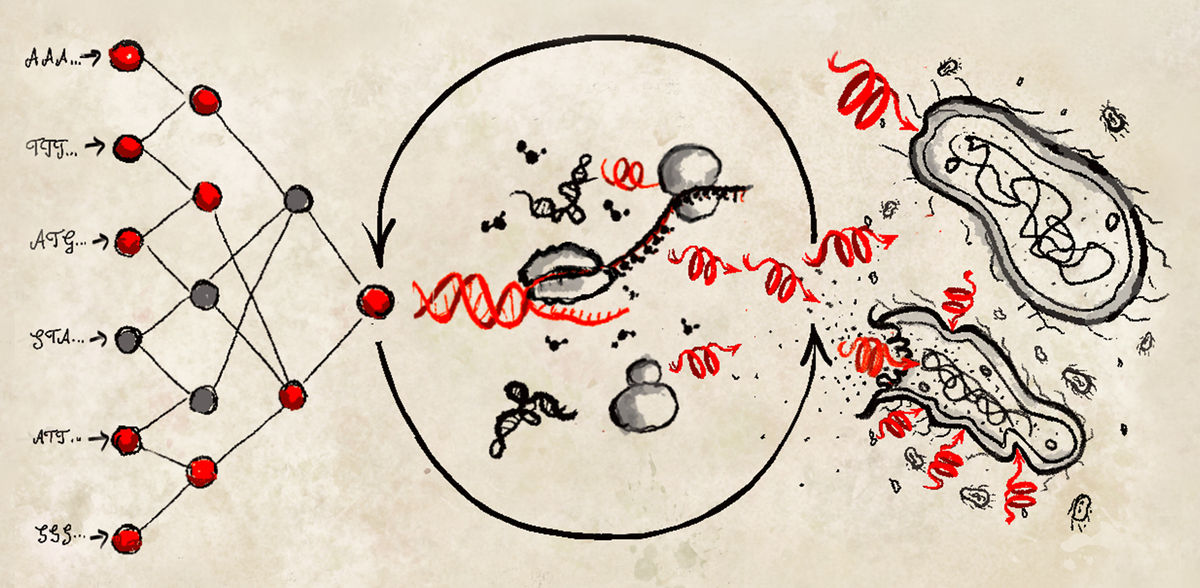
"Dans l'apprentissage profond, un réseau neuronal - des algorithmes inspirés du cerveau humain - apprend à partir de grandes quantités de données. Ce type d'apprentissage automatique est très prometteur pour la découverte de peptides et la conception de novo. Cependant, il est généralement suivi de la synthèse chimique de peptides pour la validation expérimentale, ce qui est assez difficile et prend du temps et limite considérablement le nombre de peptides qui peuvent être synthétisés chimiquement", explique Amir Pandi, auteur principal de l'étude.
Pour surmonter ces limitations, l'équipe de recherche a mis en place un pipeline de synthèse protéique acellulaire (CFPS) pour la production rapide et rentable de peptides antimicrobiens (AMP) directement à partir de modèles d'ADN. Le nouveau protocole fournit une méthode rapide, peu coûteuse et à haut débit pour le criblage des peptides antimicrobiens.
L'équipe a d'abord utilisé l'apprentissage profond génératif pour concevoir des milliers de PGA de novo, puis l'apprentissage profond prédictif pour les réduire à 500 candidats. Parmi ces derniers, le criblage avec le pipeline acellulaire a permis d'identifier 30 AMP fonctionnels, que les chercheurs ont ensuite caractérisés par des simulations de dynamique moléculaire, une activité antimicrobienne et une toxicité.
Notamment, six des AMP de novo ont présenté une activité à large spectre contre des pathogènes multirésistants et n'ont pas développé de résistance bactérienne.
"Nous avons grandement bénéficié de la combinaison de la biologie synthétique acellulaire, de l'intelligence artificielle et d'une approche à haut débit. En augmentant le nombre de candidats pouvant être testés expérimentalement en moins de 24 heures, les chances de trouver des AMP actifs ont augmenté", explique Amir Pandi. "Ainsi, notre pipeline CFPS ne se contente pas de compléter les récentes avancées en matière de conception computationnelle. Il permet également d'explorer plus rapidement et à moindre coût la relation entre la conception et la fonction des peptides bioactifs." Tobias Erb ajoute : "Cette nouvelle méthode à l'interface de la biologie synthétique et de l'apprentissage automatique intéressera les scientifiques travaillant dans les domaines de la biomédecine et des peptides bioactifs."
Les prochaines étapes consistent à améliorer encore le rendement de la production de peptides ainsi qu'à utiliser l'IA et les approches de la biologie synthétique pour concevoir de nouveaux AMP plus stables, moins toxiques ou ajoutant un mode d'action spécifique. Les chercheurs prévoient également d'appliquer des modèles génératifs profonds augmentés dans lesquels la machine apprend des représentations moléculaires pour les propriétés souhaitées, ce qui améliorerait le taux de réussite de l'identification des candidats actifs.
Note: Cet article a été traduit à l'aide d'un système informatique sans intervention humaine. LUMITOS propose ces traductions automatiques pour présenter un plus large éventail d'actualités. Comme cet article a été traduit avec traduction automatique, il est possible qu'il contienne des erreurs de vocabulaire, de syntaxe ou de grammaire. L'article original dans Anglais peut être trouvé ici.