Wie Enzyme Zuckerbäume bauen
Erkenntnisse könnten Entwicklung neuer proteinbasierter Medikamente beschleunigen
Anzeigen
Forscher klärten erstmals mithilfe der Kryo-Elektronenmikroskopie die Struktur und Funktion eines kleinen, in Zellmembranen steckenden Enzyms auf. Dieses baut komplexe Strukturen aus Zuckermolekülen auf. Die Erkenntnisse könnten die Entwicklung neuer proteinbasierter Medikamente beschleunigen.

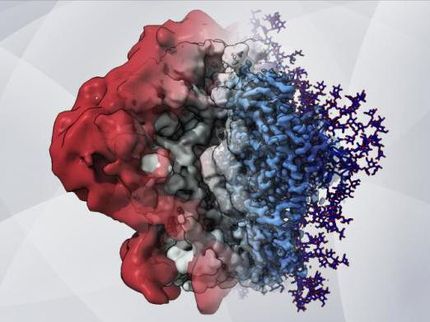
Das Enzym ALG6 besteht aus einem strukturell konservierten (grün) und einem strukturell variablen (rot) Modul. Der Antikörper, der an das Enzym bindet (lila, türkis), ermöglichte dessen Analyse mittels Kryo-EM.
ETH Zürich / Joël Bloch
Viele Membranproteine werden in Zellen höherer Lebewesen mit komplexen Strukturen aus Zuckermolekülen, sogenannten Glykanen, bestückt. Diese molekularen Zuckerbäumchen sind nicht nur äusserst vielfältig, sondern auch charakteristisch für den jeweiligen Organismus, einen Zelltyp oder dessen Differenzierunsgrad. Unterschiedliche Glykane sind zum Beispiel auch der Schlüssel zu den unterschiedlichen Blutgruppen beim Menschen.
Eine besondere Klasse der Glykane sind mit Lipiden verknüpfte Zuckermoleküle: Lipid-gebundene Oligosaccharide. Diese bestehen aus einem Fettmolekül, das in der Zellmembran verankert ist, und einem Zuckerteil, der entweder ins Innere von Zellorganellen oder in den Zellaussenraum ragt.
Forscher der ETH Zürich, der Universität Bern sowie der Universität Chicago haben nun die Struktur eines der Enzyme aufgeklärt, welche für den Aufbau der Lipid-gebundenen Oligosaccharide verantwortlich sind. Die entsprechende Studie ist in der jüngsten Ausgabe des Fachmagazins Nature erschienen.
Modulare Architektur von Proteinen
Das gefundene Enzym namens ALG6 ist Mitglied einer ganzen Überfamilie von Enzymen, welche die Forscher als Glykosyltransferasen der Kategorie C bezeichnen. Sie sitzen in Zellmembranen und koppeln einfachere Zuckermoleküle an andere Zucker, um Zuckerbäumchen aufzubauen. Sie verknüpfen zudem Zuckermoleküle und sogar ganze Zuckerbäumchen mit Proteinen. Damit übernimmt diese Enzymfamilie mehrere biologische Schlüsselrollen, und zwar in allen möglichen Organismen von Bakterien über Pilze bis hin zu hochentwickelten Säugetieren.
Für Biologen war diese Enzymüberfamilie lange ein Rätsel, da die Strukturen der einzelnen Familienmitglieder kaum Gemeinsamkeiten aufweisen. Der einzige gemeinsame Nenner war, dass es sich um Membranproteine handelt, welche Zucker von einem Molekül auf ein anderes transferieren und dass die für den Transfer verwendeten Zucker immer an Lipide geknüpft sind.
Mit Hilfe der Struktur von ALG6 fand die von ETH-Professor Kaspar Locher angeführte Gruppe nun heraus, dass die Mitglieder dieser Enzymfamilie modular aufgebaut sind. Es scheint, als ob die ALG6 und seine Verwandten aus einem Modul, dessen Struktur in der Entwicklungsgeschichte erhalten blieb, sowie aus einem zweiten strukturell variablen Modul bestehen.
«Wir vermuten, dass es der modulare Aufbau den Enzymen ermöglichte, sich in verschiedene Richtungen zu entwickeln und somit ein weites Spektrum an verschiedenen Substraten zu erschliessen und umzusetzen», sagt Joël Bloch, Doktorand in Lochers Gruppe und Erstautor der Studie.
Die Forschungsergebnisse erklären erstmals den Mechanismus dieser Enzymfamilie. «Dies hat weitreichende Auswirkungen für die Zellbiologie sowie für die Herstellung therapeutischer Substanzen, die auf der Zuckerbiologie beruhen», so der ETH-Doktorand. Insbesondere die Herstellung von massgeschneiderten therapeutischen Antikörpern, an denen die Pharmaindustrie stark interessiert ist, könnten von den neuen Erkenntnissen profitieren.
Rekord in der Kryo-Elektronenmikroskopie
Die Forscher sehen ihre Resultate zudem als Durchbruch in der Aufklärung von Molekülstrukturen von Proteinen mittels Kryo-Elektronenmikroskopie (Kryo-EM). Die bahnbrechende Technologie, für welche 2017 der Chemienobelpreis unter anderen an den Waadtländer Jacques Dubochet vergeben wurde, hat sich zur wichtigsten Methode bei der Strukturaufklärung von grossen Molekülkomplexen gemausert. Die hochauflösende Strukturaufklärung kleiner Proteine, insbesondere von solchen, die in Membranen eingebettet sind, gelang damit hingegen nicht. Der Grund: die Mikroskopie-Daten von kleinen gemessenen Partikeln verunmöglichen präzise strukturelle Berechnungen in der Strukturaufklärung.
Die ETH-Forscher haben dieses Problem in Zusammenarbeit mit einer Forschungsgruppe der Universität Chicago nun lösen können. In Zusammenarbeit mit den dortigen Forschern stellten sie einen synthetischen Antikörper her, welcher an ALG6 bindet. Dieser Antikörper erhöhte das Gewicht des Enzyms ALG6 so, dass dessen Struktur mithilfe der Kryo-EM hochaufgelöst aufgeklärt werden konnte.
«Die auf diese Weise erzielte Auflösung ist aktuell der Weltrekord bei der Strukturauflösung eines membrangebundenen Komplexes dieser Grösse», sagt Kaspar Locher nicht ohne Stolz. Diese Weiterentwicklung der Kryo-EM werde es vielen anderen Wissenschaftlern ermöglichen, Strukturen von kleinen Membranproteinen aufzuklären. «Dadurch wird die Wissenschaft rasch Fortschritte erzielen bei der Untersuchung von Membranproteinen, die mit einer Vielzahl von Krankheiten in Verbindung stehen», betont der ETH-Professor.
Ein chemo-enzymatischer Baukasten
Damit nicht genug: In Zusammenarbeit mit Chemikern der Universität Bern entwickelten die ETH-Forscher Synthesemethoden, mit denen sie hochkomplexe Lipid-gebundene Oligosaccharide im Labor herstellen konnten. Mit herkömmlichen Synthesen der organischen Chemie war dies bisher nicht möglich.
Dies wiederum bot den Forschern neue Einblicke in den essenziellen Stoffwechselweg der Biosynthese von Lipid-gebundenen Oligosacchariden. Die Wissenschaftler können nun besser erklären, wie Zellen komplexe Glykane herstellen. «Dies ist ein Meilenstein in der Glykobiologie, der künftig für die Herstellung von Zucker-Protein-Verbindungen bedeutend sein könnte», sagt Locher.