L'étude la plus approfondie du génome humain
Une nouvelle ressource pour la recherche génomique dans le monde entier
En 2003, le projet du génome humain (HGP) a réussi à cartographier pour la première fois plus de 90 % du génome humain. Par la suite, le projet 1 000 génomes (2007 - 2015), qui a rassemblé les données de plus de 2 500 personnes de populations et de sexes différents, a permis d'obtenir un premier aperçu de la diversité du génome humain. Aujourd'hui, dix ans après la fin du projet, un consortium de recherche international a réanalysé les échantillons à l'aide de techniques de pointe, en collaboration avec le professeur Tobias Marschall, bioinformaticien à l'université Heinrich Heine de Düsseldorf (HHU). Les résultats de cette analyse sont présentés dans deux publications parues dans le même numéro de la revue scientifique Nature. Ils montrent la diversité du génome humain à un niveau de profondeur sans précédent.
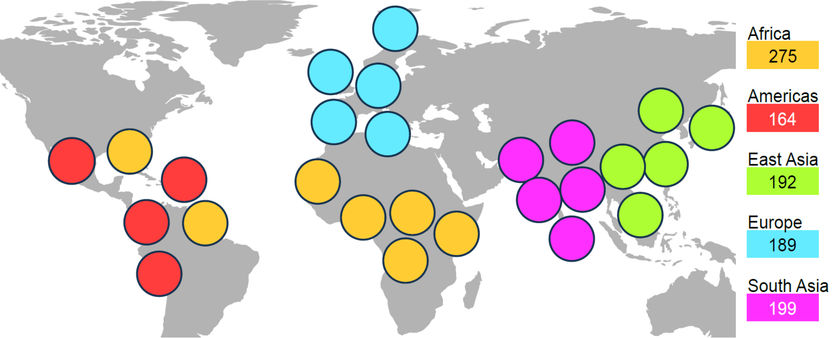
Les études ont analysé des ensembles de données génomiques provenant de cinq continents et de 26 populations. Il a ainsi été possible de cartographier une diversité particulièrement importante du génome humain.
Copyright: Siegfried Schloissnig / HHU – Berit Meisenkothen
S'appuyant sur le projet HGP, le projet 1 000 génomes avait pour objectif de séquencer le génome humain d'un échantillon représentatif de la population mondiale. À la fin du projet en 2015, l'objectif avait été dépassé et les données de 2 500 personnes des cinq continents et de 26 populations avaient été rassemblées. Les deux projets ont contribué de manière significative à la compréhension du génome humain.
Dix ans après la fin du projet, un groupe de recherche international auquel participe l'équipe dirigée par le professeur Marschall (Institut de biométrie médicale et de bio-informatique) a examiné le génome humain plus en détail. Dans les études publiées dans la revue scientifique Nature, les génomes collectés dans le cadre du projet 1 000 génomes ont été réanalysés à l'aide de technologies avancées, qui n'étaient pas disponibles en 2015.
La nouveauté : À l'époque du projet 1 000 génomes, le séquençage, c'est-à-dire la détermination de la séquence des lettres du génome humain, reposait en grande partie sur de courts segments d'ADN, qui n'étaient pas suffisants pour assembler un génome complet. Les nouvelles méthodes de séquençage dites "long-read" permettent désormais une analyse beaucoup plus détaillée des génomes. Ces technologies fournissent les séquences de segments d'ADN plus longs en un seul morceau, ce qui facilite l'identification des différences génétiques entre les individus.
Ces "variantes génétiques" peuvent se présenter sous diverses formes, telles que des différences d'une ou plusieurs paires de bases - les lettres - de la séquence d'ADN. Cependant, ils peuvent aussi être plus étendus, par exemple lorsque de longs segments d'ADN sont supprimés, inversés, répétés ou ajoutés chez certains individus. Ces cas sont également appelés variantes structurelles et jouent un rôle important dans le développement d'une variété de maladies génétiques, y compris des syndromes génétiques rares et encore inexpliqués.
Le pangénome : La cartographie du génome humain
En 2023, le Human Pangenome Reference Project (HPRC), auquel le professeur Marschall a également participé, a publié un projet de "pangénome de référence", c'est-à-dire une carte de la diversité génétique humaine, basée sur 47 individus. L'objectif est de remplacer à l'avenir le génome de référence utilisé jusqu'à présent par ce projet. Les données de la nouvelle étude y contribueront également.
Dans la première des études publiées (Schloissnig, Pani, et al.), 1 019 génomes ont été séquencés, ce qui rend cette cohorte plus de 20 fois plus importante que les données du HPRC. Ce nouvel ensemble de données de référence, nettement plus important, s'avère particulièrement utile pour l'étude des variantes structurelles qui se produisent moins fréquemment dans la population. "Il est essentiel de disposer de variantes dans une cohorte diversifiée d'individus sains pour mieux comprendre quelles variantes dans les génomes des patients peuvent être à l'origine des affections dont ils souffrent", explique le professeur Dr Dagmar Wieczorek (Institut de génétique humaine de l'université de Humboldt), qui a également participé à l'étude.
La seconde étude (Logsdon, Ebert, Audano, Loftus, et al.) élargit également les connaissances actuelles sur le génome humain. Toutefois, l'accent n'a pas été mis sur le nombre de génomes, mais plutôt sur le séquençage le plus complet possible des génomes. 65 échantillons, qui font également partie du projet 1 000 génomes, ont été examinés à l'aide de méthodes de séquençage très sophistiquées. Les chercheurs ont pu reconstruire des séquences génomiques complètes (appelées "T2T" ou "télomère à télomère") pour 1 161 chromosomes (39 %). "Ce résultat est particulièrement remarquable, car les chromosomes humains peuvent contenir des centaines de millions de paires de bases et il n'y a que quelques années que la première reconstruction complète d'un génome individuel a été réalisée", explique le professeur Alexander Dilthey, bioinformaticien à l'HHU (chef de l'équipe de recherche à l'Institut de microbiologie médicale et d'hygiène hospitalière), qui a également participé à l'étude.
En outre, les génomes complets permettent désormais de comprendre certaines régions, comme les centromères, qui n'étaient pas accessibles par les méthodes conventionnelles. Les centromères sont les points où les deux chromatides sont reliées lors de la division cellulaire - ils forment le X bien connu. Les recherches sur l'importance et les conséquences des variantes génétiques dans les centromères ont été limitées jusqu'à présent. La nouvelle étude permet désormais de poursuivre l'étude de leur influence, par exemple sur les troubles immunitaires et les cancers.
Le professeur Jan Korbel, du Laboratoire européen de biologie moléculaire (EMBL) à Heidelberg, coauteur des deux articles, estime que la publication simultanée des deux études est une réussite particulière : "Bien que la première étude utilise des méthodes de séquençage moins sophistiquées, elle est basée sur une cohorte beaucoup plus importante, tandis que la seconde étude est basée sur une cohorte plus petite mais utilise des méthodes de séquençage plus avancées. Cela nous permet d'obtenir des informations extrêmement solides et précises sur les variations de nos génomes".
Une nouvelle ressource pour la recherche sur le génome dans le monde entier
Le professeur Marschall souligne également que les résultats des deux études ne fournissent pas seulement des informations importantes, mais qu'ils augmentent également de manière significative la quantité de données disponibles, ce qui aura un impact positif sur la recherche à long terme. "Ces études constituent une ressource importante et pertinente sur le plan médical, qui peut désormais être utilisée par les chercheurs du monde entier pour mieux comprendre les mécanismes mutationnels à l'origine des variations du génome humain", déclare le professeur Marschall. "Il s'agit d'un exemple remarquable de recherche collaborative et de science ouverte, qui ouvre de nouvelles perspectives dans la recherche sur le génome et représente une étape vers une connaissance plus complète du génome humain. Je suis convaincu que, sur la base de ces résultats importants, nous serons en mesure d'identifier de nombreux liens entre les variantes génétiques structurelles et les risques de maladie à l'avenir."
Les nouveaux ensembles de données ont été mis à la disposition des chercheurs du monde entier pour qu'ils les analysent et les utilisent.
Note: Cet article a été traduit à l'aide d'un système informatique sans intervention humaine. LUMITOS propose ces traductions automatiques pour présenter un plus large éventail d'actualités. Comme cet article a été traduit avec traduction automatique, il est possible qu'il contienne des erreurs de vocabulaire, de syntaxe ou de grammaire. L'article original dans Anglais peut être trouvé ici.
Publication originale
Siegfried Schloissnig, Samarendra Pani, Jana Ebler, Carsten Hain, Vasiliki Tsapalou, Arda Söylev, Patrick Hüther, Hufsah Ashraf, Timofey Prodanov, Mila Asparuhova, ...Tobias Rausch, Tobias Marschall, Jan O. Korbel; "Structural variation in 1,019 diverse humans based on long-read sequencing"; Nature, 2025-7-23
Glennis A. Logsdon, Peter Ebert, Peter A. Audano, Mark Loftus, David Porubsky, Jana Ebler, Feyza Yilmaz, Pille Hallast, Timofey Prodanov, DongAhn Yoo, Carolyn A. Paisie, William T. Harvey, Xuefang Zhao, Gianni V. Martino, Mir Henglin, ...Miriam K. Konkel, Jan O. Korbel, Charles Lee, Christine R. Beck, Evan E. Eichler, Tobias Marschall; "Complex genetic variation in nearly complete human genomes"; Nature, 2025-7-23