Ver la propagación del cáncer a través de una ventana computacional
Un modelo computacional permite a los investigadores simular interacciones a escala celular a distancias sin precedentes en la vasculatura humana
Ingenieros biomédicos de la Universidad de Duke han mejorado notablemente las capacidades de un modelo computacional que simula el movimiento de células cancerosas individuales a través de largas distancias en todo el cuerpo humano.
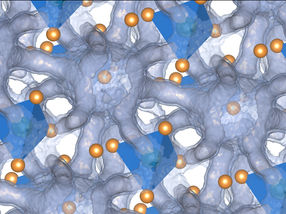
Representación artística de cómo un nuevo sistema de modelización computacional simula el desplazamiento de una célula cancerosa por el cuerpo humano. Para tener en cuenta millones de interacciones celulares, sólo crea simulaciones detalladas de las células situadas en las inmediaciones de la célula cancerosa mientras circula por el torrente sanguíneo.
Duke University
El método, denominado "Adaptive Physics Refinement (APR)", capta con detalle las interacciones celulares y sus efectos en la trayectoria de las células, lo que ofrece información muy valiosa sobre los viajes de las células cancerosas metastásicas.
"En el torrente sanguíneo, las células cancerosas se ven influidas por el choque y el movimiento de los glóbulos rojos cercanos y otras interacciones celulares", explica Aristóteles Martín, doctorando del laboratorio Amanda Randles de Ingeniería Biomédica de Duke. "Pero es imposible simular el movimiento de todos los glóbulos rojos en todos los vasos sanguíneos del cuerpo, así que tuvimos que encontrar una forma de sortear esas limitaciones computacionales".
La investigación se presentó el 15 de noviembre de 2023 en la Conferencia Internacional de Computación, Redes, Almacenamiento y Análisis de Alto Rendimiento (SC23). Esta conferencia es la principal reunión internacional en el campo de la computación de alto rendimiento, patrocinada por ACM e IEEE-CS.
Descifrar la dinámica de navegación de las células cancerosas a través de los vasos sanguíneos del organismo sigue siendo una cuestión crítica y compleja en los estudios sobre el cáncer, crucial para su detección precoz y un posible tratamiento dirigido. Estudiar estos procesos en pacientes vivos, sin embargo, no es factible, y en su lugar requiere modelos computacionales avanzados para simular la dinámica de las células cancerosas.
Amanda Randles, catedrática asociada Alfred Winborne y Victoria Stover Mordecai de Ciencias Biomédicas en Duke, lleva más de una década creando y avanzando métodos computacionales que exploran estos procesos fundamentales. Una de sus contribuciones más notables es HARVEY, un paquete de simulación hemodinámica altamente escalable diseñado para funcionar en los superordenadores más avanzados del mundo.
Pero incluso los superordenadores tienen sus límites.
Para calcular la trayectoria de una sola célula cancerosa, los modelos deben captar sus interacciones microscópicas con los glóbulos rojos circundantes. Sin embargo, el cuerpo humano contiene unos 25 billones de glóbulos rojos y cinco litros de sangre. Con los mayores superordenadores actuales, los modelos más avanzados sólo pueden recrear una región que contenga el uno por ciento de este volumen a resolución celular, un dominio limitado que aún incluye varios cientos de millones de glóbulos rojos.
Para eludir este problema, un amplio equipo con colaboradores del Laboratorio Nacional Lawrence Livermore (LLNL) y el Laboratorio Nacional Oak Ridge (ORNL) dirigido por Sayan Roychowdhury, antiguo estudiante de doctorado en el laboratorio de Randles, ha adoptado un nuevo enfoque. Ampliando el algoritmo existente en el laboratorio para incluir las interacciones con millones de glóbulos rojos vecinos, APR crea una ventana de alta resolución que rastrea la célula de interés a medida que se desplaza por la vasculatura.
Parte del truco para que este enfoque funcione consiste en acoplar la ventana móvil a una simulación de todo el dominio vascular, modelando la sangre como un fluido a granel. Este enfoque es análogo a la simulación del comportamiento de un barco de juguete mientras flota en una corriente. El enfoque más sencillo sería modelar todo el curso de agua a la máxima resolución de principio a fin. Sin embargo, en tal escenario, la gran mayoría de los recursos computacionales capturarían precisamente fenómenos físicos que ocurren lejos del barco, lo que sería poco práctico en el mejor de los casos.
En su lugar, es mucho más eficiente modelar la región cercana al barco con la mayor precisión posible mientras se modela el resto de la corriente con una resolución más modesta. A medida que la embarcación se acerca a elementos complejos como rocas, remolinos y rápidos, el modelo los capta con precisión una vez que están lo suficientemente cerca como para tener un impacto mensurable en la trayectoria de la embarcación. El resultado es una simulación precisa y mucho más eficaz, que permite explorar tramos de río mucho más largos.
"El mayor número de células que hemos simulado a la vez es de 580 millones", explica Samreen Mahmud, estudiante de doctorado del laboratorio Randles. "Nuestro objetivo era maximizar el tamaño de la ventana para ver cuántas celdas podíamos capturar utilizando un superordenador de clase dirigente. Después nos centramos en reducir el coste computacional y trasladar el método a la nube de forma eficiente."
La eficacia del algoritmo en la realización de simulaciones a gran escala se puso de manifiesto en el estudio al simular el tránsito de una célula cancerosa a través de un centímetro utilizando un único nodo en los servicios web de Amazon (AWS) durante 500 horas. Al emplear Adaptive Physics Refinement, la demanda de memoria computacional se redujo drásticamente de petabytes a gigabytes más manejables.
Según los investigadores, los resultados podrían cambiar las reglas del juego para otros laboratorios que estudian el cáncer o desarrollan dispositivos biomédicos. Podría, por ejemplo, ayudar a los investigadores a entender el aspecto mecánico y físico de la metástasis del cáncer aislando factores que serían difíciles o incluso imposibles de hacer dentro de un montaje experimental. También representa un salto significativo en las capacidades de computación de alto rendimiento, ya que facilita la aplicación práctica del modelado de grandes cantidades de glóbulos rojos con capacidad limitada.
De cara al futuro, el equipo tiene previsto seguir añadiendo funciones a su software de modelización, como la adhesión entre células y los cambios en el comportamiento celular cerca de las paredes de los vasos sanguíneos. También quieren investigar cómo se mueven los grupos de células cancerosas por la vasculatura, ya que los estudios clínicos han demostrado que viajar en grupos aumenta el potencial de las células metastásicas para formar nuevos tumores.
"Esperamos que métodos como APR ayuden a democratizar el modelado computacional a escala celular nivelando el campo de juego", dijo Randles. "Queremos permitir a los investigadores sin acceso a los mayores superordenadores del mundo utilizar enfoques computacionales para estudiar la dinámica del cáncer."
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.
Publicación original
Sayan Roychowdhury, Samreen T. Mahmud, Aristotle Martin, Peter Balogh, Daniel F. Puleri, John Gounley, Erik W. Draeger, Amanda Randles; "Enhancing Adaptive Physics Refinement Simulations Through the Addition of Realistic Red Blood Cell Counts"; Proceedings of the International Conference for High Performance Computing, Networking, Storage and Analysis, 2023-11-11