Voir la propagation du cancer à travers une fenêtre de calcul
Un modèle informatique permet aux chercheurs de simuler les interactions à l'échelle cellulaire sur des distances sans précédent dans le système vasculaire humain
Des ingénieurs biomédicaux de l'université Duke ont considérablement amélioré les capacités d'un modèle informatique qui simule le mouvement de cellules cancéreuses individuelles sur de longues distances à l'intérieur du corps humain.
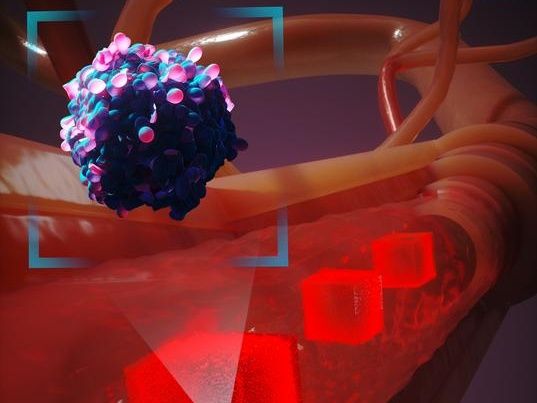
Une représentation artistique de la manière dont un nouveau système de modélisation informatique simule une cellule cancéreuse se déplaçant dans le corps humain. Pour tenir compte des millions d'interactions cellulaires, le système ne crée que des simulations détaillées des cellules situées à proximité immédiate de la cellule cancéreuse lorsqu'elle circule dans le flux sanguin.
Duke University
Baptisée "Adaptive Physics Refinement (APR)", cette approche permet de saisir les interactions cellulaires détaillées et leurs effets sur la trajectoire des cellules, offrant ainsi des informations précieuses sur les déplacements des cellules cancéreuses métastatiques.
"Les cellules cancéreuses dans notre circulation sanguine sont influencées par les chocs avec les globules rouges voisins et par d'autres interactions cellulaires", explique Aristote Martin, candidat au doctorat dans le laboratoire d'Amanda Randles à Duke Biomedical Engineering. "Mais il est impossible de simuler le mouvement de chaque globule rouge dans tous les vaisseaux sanguins du corps, et nous avons donc dû trouver un moyen de contourner ces limitations informatiques.
La recherche a été présentée le 15 novembre 2023 lors de la conférence internationale sur le calcul de haute performance, la mise en réseau, le stockage et l'analyse (SC23). Cette conférence est la première réunion internationale dans le domaine du calcul de haute performance, parrainée par ACM et IEEE-CS.
Le décryptage de la dynamique de navigation des cellules cancéreuses dans les vaisseaux sanguins de l'organisme reste une question critique et complexe dans les études sur le cancer, cruciale pour la détection précoce et le traitement ciblé potentiel. L'étude de ces processus sur des patients vivants n'est toutefois pas réalisable et nécessite des modèles informatiques avancés pour simuler la dynamique des cellules cancéreuses.
Amanda Randles, professeur agrégé de sciences biomédicales à Duke (Alfred Winborne and Victoria Stover Mordecai Associate Professor of Biomedical Sciences), crée et fait progresser les méthodes informatiques qui explorent ces processus fondamentaux depuis plus d'une décennie. L'une de ses contributions notables est HARVEY, un logiciel de simulation hémodynamique hautement évolutif conçu pour fonctionner sur les superordinateurs les plus avancés du monde.
Mais même les superordinateurs ont leurs limites.
Pour calculer la trajectoire d'une seule cellule cancéreuse, les modèles doivent tenir compte de ses interactions microscopiques avec les globules rouges environnants. Or, le corps humain contient environ 25 000 milliards de globules rouges et cinq litres de sang. En utilisant les plus grands superordinateurs actuels, les modèles de pointe ne peuvent recréer qu'une région contenant un pour cent de ce volume à une résolution cellulaire - un domaine limité qui comprend encore plusieurs centaines de millions de globules rouges.
Pour contourner ce problème, une grande équipe de collaborateurs du Lawrence Livermore National Laboratory (LLNL) et du Oak Ridge National Laboratory (ORNL), dirigée par Sayan Roychowdhury, ancien doctorant du laboratoire Randles, a adopté une nouvelle approche. En étendant l'algorithme existant du laboratoire pour inclure les interactions avec des millions de globules rouges voisins, l'APR crée une fenêtre à haute résolution qui suit la cellule d'intérêt au fur et à mesure qu'elle se déplace dans le système vasculaire.
Pour que cette approche fonctionne, il faut coupler la fenêtre mobile à une simulation de l'ensemble du domaine vasculaire, en modélisant le sang comme un fluide en vrac. Cette approche est analogue à la simulation du comportement d'un petit bateau flottant sur un cours d'eau. L'approche la plus simple consisterait à modéliser l'ensemble de la voie d'eau à la résolution la plus élevée, du début à la fin. Cependant, dans un tel scénario, la grande majorité des ressources informatiques devraient capturer précisément les phénomènes physiques qui se produisent loin du bateau, ce qui ne serait pas pratique dans le meilleur des cas.
Il est bien plus efficace de modéliser la région proche du bateau aussi précisément que possible, tout en modélisant le reste du cours d'eau à une résolution plus modeste. Lorsque le bateau s'approche d'éléments complexes tels que des rochers, des tourbillons et des rapides, le modèle les saisit avec précision dès qu'ils sont suffisamment proches pour avoir un impact mesurable sur la trajectoire du bateau. Il en résulte une simulation précise et beaucoup plus efficace, qui permet d'explorer des tronçons de rivière beaucoup plus longs.
"Le plus grand nombre de cellules que nous ayons jamais simulées en une seule fois est de 580 millions", explique Samreen Mahmud, étudiante en doctorat au Randles Lab. "Notre objectif était de maximiser la taille de la fenêtre pour voir combien de cellules nous pouvions capturer en utilisant un superordinateur de premier plan. Nous nous sommes ensuite attachés à réduire les coûts de calcul et à transférer efficacement la méthode dans le nuage."
L'efficacité de l'algorithme dans la réalisation de simulations à grande échelle a été démontrée dans l'étude en simulant le transit d'une cellule cancéreuse sur un centimètre à l'aide d'un seul nœud sur les services Web d'Amazon (AWS) pendant 500 heures. L'utilisation de l'affinement adaptatif de la physique a permis de réduire considérablement la demande de mémoire de calcul, qui est passée de pétaoctets à des gigaoctets plus faciles à gérer.
Selon les chercheurs, ces résultats pourraient changer la donne pour d'autres laboratoires qui étudient le cancer ou développent des appareils biomédicaux. Ils pourraient, par exemple, aider les chercheurs à comprendre l'aspect mécanique et physique des métastases cancéreuses en isolant des facteurs qu'il serait difficile, voire impossible, d'isoler dans le cadre d'un dispositif expérimental. Il s'agit également d'une avancée significative dans les capacités de calcul à haute performance, qui facilite l'application pratique de la modélisation de vastes quantités de globules rouges avec une capacité limitée.
À l'avenir, l'équipe prévoit de continuer à ajouter des fonctionnalités à son logiciel de modélisation, telles que l'adhésion entre les cellules et les changements de comportement cellulaire à proximité des parois des vaisseaux sanguins. Elle souhaite également étudier la manière dont les groupes de cellules cancéreuses se déplacent dans le système vasculaire, car des études cliniques ont montré que le fait de se déplacer en groupe augmente le potentiel des cellules métastatiques à former de nouvelles tumeurs.
"Nous espérons que des méthodes telles que l'APR contribueront à démocratiser la modélisation informatique à l'échelle cellulaire en uniformisant les règles du jeu", a déclaré M. Randles. "Nous voulons permettre aux chercheurs qui n'ont pas accès aux plus grands superordinateurs du monde d'utiliser des approches informatiques pour étudier la dynamique du cancer."
Note: Cet article a été traduit à l'aide d'un système informatique sans intervention humaine. LUMITOS propose ces traductions automatiques pour présenter un plus large éventail d'actualités. Comme cet article a été traduit avec traduction automatique, il est possible qu'il contienne des erreurs de vocabulaire, de syntaxe ou de grammaire. L'article original dans Anglais peut être trouvé ici.
Publication originale
Sayan Roychowdhury, Samreen T. Mahmud, Aristotle Martin, Peter Balogh, Daniel F. Puleri, John Gounley, Erik W. Draeger, Amanda Randles; "Enhancing Adaptive Physics Refinement Simulations Through the Addition of Realistic Red Blood Cell Counts"; Proceedings of the International Conference for High Performance Computing, Networking, Storage and Analysis, 2023-11-11