Wirkmechanismus neuer Antibiotika gegen Tuberkulose entschlüsselt
Strukturelle Analyse des Proteins DprE1 in PNAS veröffentlicht
Anzeigen
Die Tuberkulose gehört zu den weltweit häufigsten Infektionskrankheiten. Zunehmend resistente Bakterien erschweren die Behandlung und Eindämmung der Infektionen. Biotechnologen aus der Universität Birmingham und dem Forschungszentrum Jülich ist es erstmals gelungen, die Struktur des Proteins DprE1 zu entschlüsseln, das für das Überleben von Mycobacterium tuberculosis, dem häufigsten Erreger, essenziell ist. Die in der Fachzeitschrift PNAS veröffentlichte Arbeit zeigt den Wirkmechanismus neuer, hochpotenter Antibiotika und liefert einen Ansatzpunkt für das Design neuer Wirkstoffe.

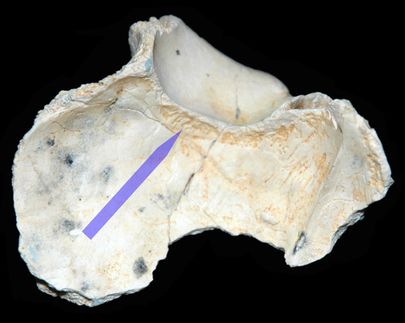
Neuer Wirkstoff CT319 (violette Kugeln) dockt an der molekularen Struktur des Proteins DprE1 an.
Quelle: Dr. K. Futterer, University of Birmingham
Schätzungen der Weltgesundheitsorganisation WHO zufolge ist etwa ein Drittel der Weltbevölkerung mit Mycobacterium tuberculosis infiziert. Rund jeder Zehnte davon erkrankt im Laufe seines Lebens an Tuberkulose, wobei sich das Risiko für HIV-Infizierte auf das über 20-fache erhöht. Das Enyzm DprE1 spielt eine entscheidende Rolle beim Aufbau der Zellwand des Bakteriums. Erst vor einigen Jahren wiesen Forscher nach, dass es für die Produktion von Arabinose zuständig ist. Der Zuckerbaustein ist Bestandteil der mehrschichtigen Zellwand der Bakterien. Ohne ihn kann der Tuberkulose-Erreger nicht überleben.
„Seit zwei Jahren schon haben wir versucht, das Protein in die Hände zu bekommen“, freut sich der Dr. Lothar Eggeling vom Institut für Jülicher Bio-und Geowissenschaften, Bereich Biotechnologie. „Da das Enzym DprE1 nur in Mycobakterien und verwandten Organismen, nicht aber im Menschen vorkommt, eignet es sich hervorragend als Wirkort für Antibiotika. Ähnlich ist es beim alten, erprobten Penicillin, das ja auch in die Zellwandsynthese von Bakterien eingreift.“ Die Jülicher Wissenschaftler haben das Protein isoliert und Kollegen der School of Biosciences an der Universität Birmingham, mit denen sie schon seit Jahren erfolgreich auf dem Gebiet der Zellwandsynthese zusammenarbeiten, zur weiteren Analyse zur Verfügung gestellt.
In ihrem Fachartikel in PNAS zeigen die Wissenschaftler, wie erst seit wenigen Jahren eingesetzte, hochwirksame Antibiotika aus der Gruppe der Benzothiazinone wirken. Sie docken auf molekularer Ebene an der Aminosäure Cys387, einem Bestandteil des Enzyms, an und unterdrücken dessen Funktion bei der Herstellung von Arabinose. „Unerwartet war, dass das Enzym neben dem schon bekannten Ausgangswirkstoff CT325 auch noch durch eine sehr ähnliche Substanz, CT319, gehemmt wird. Es scheint, dass dabei eine andere Region im Enyzm, die als loop 316-322 bekannt ist, eine Rolle spielt“, so Eggeling. „Das erweitert die Möglichkeiten, neue Wirkstoffe zu finden, noch einmal erheblich.“
Originalveröffentlichung
Sarah M. Batt et al.; Structural basis of inhibition of Mycobacterium tuberculosis DprE1 by benzothiazinone inhibitors; PNAS (Published online before print June 25)