Mini-organi coltivati rivelano le armi dei batteri aggressivi
Lo studio apre la strada all'utilizzo di mini-organi umani in coltura per studiare un'ampia gamma di altre infezioni gravi
Annunci
Grazie a mini-intestini cresciuti in laboratorio, i ricercatori dell'Helmholtz Institute for RNA-based Infection Research (HIRI) di Würzburg, in collaborazione con l'Università di Uppsala in Svezia, sono riusciti a mappare il modo in cui i batteri aggressivi della Shigella infettano l'intestino umano. Lo studio, pubblicato sulla rivista Nature Genetics, apre la strada all'utilizzo di mini-organi umani in coltura per studiare un'ampia gamma di altre infezioni gravi.
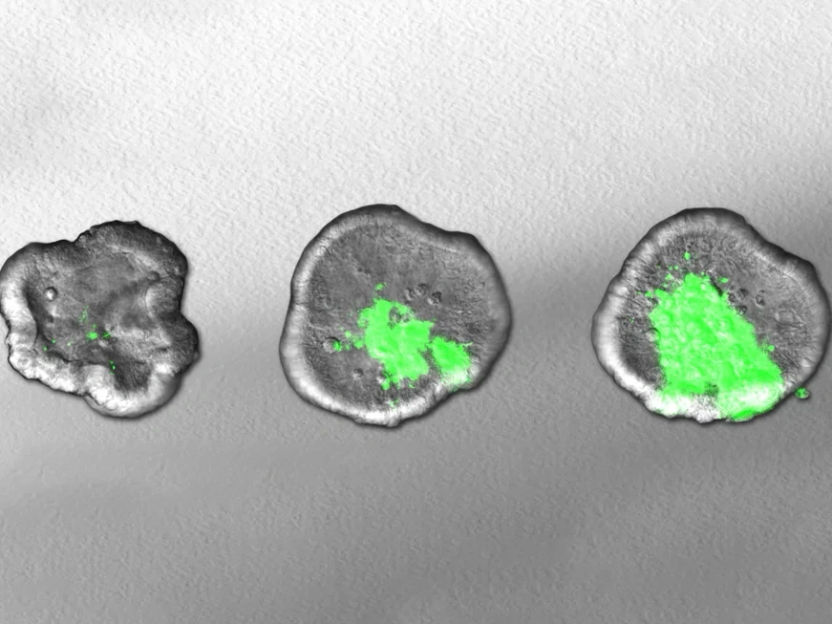
L'immagine mostra come i batteri della Shigella (in verde) si diffondono nel tempo in un intestino in miniatura cresciuto in laboratorio.
© Maria Letizia Di Martino
Capire come i batteri specifici dell'uomo ci facciano ammalare è una sfida. In un nuovo studio pubblicato su Nature Genetics, i ricercatori dimostrano che è ora possibile utilizzare mini-organi in coltura, i cosiddetti organoidi, per mappare il modo in cui i batteri colonizzano la mucosa intestinale umana. Il team dell'Istituto Helmholtz per la ricerca sulle infezioni basate sull'RNA (HIRI) di Würzburg, una sede del Centro Helmholtz di Braunschweig per la ricerca sulle infezioni (HZI) in collaborazione con la Julius-Maximilians-Universität Würzburg (JMU) e l'Università di Uppsala, si è concentrato in particolare su Shigella flexneri. Il batterio causa gravi infiammazioni intestinali nell'uomo ed è responsabile di molti decessi all'anno, soprattutto tra i bambini piccoli.
"I nostri risultati forniscono un quadro più realistico dei fattori che influenzano la colonizzazione di Shigella nel nostro intestino. Questo potrebbe consentire nuovi approcci terapeutici in futuro", afferma Lars Barquist, scienziato associato all'HIRI e professore all'Università di Toronto in Canada, nonché co-autore senior dello studio. Per la prima volta, siamo riusciti a mappare i geni di cui la Shigella ha bisogno per causare l'infezione utilizzando un modello umano che imita il tessuto intestinale". Lo studio dimostra inoltre che i mini-organi umani in coltura possono ora essere utilizzati per studiare una serie di infezioni gravi, in particolare quelle per le quali la mancanza di modelli animali di laboratorio ha precedentemente limitato la ricerca", aggiunge Maria Letizia Di Martino dell'Università di Uppsala, che ha guidato lo sviluppo del sistema sperimentale.
Modelli intestinali derivati da cellule staminali
I batteridella shigella sono agenti patogeni invasivi che attaccano i tessuti dell'organismo utilizzando una varietà di "armi" per invadere la mucosa intestinale e manipolare le funzioni del sistema immunitario del corpo. Nello studio attuale, i ricercatori si sono concentrati sull'identificazione dei geni responsabili della produzione di queste armi. Per farlo, hanno generato organoidi intestinali - modelli intestinali in miniatura - cresciuti da cellule staminali umane purificate da materiale di scarto chirurgico. Hanno poi utilizzato un metodo che elimina in modo casuale i geni batterici e hanno testato come questi cambiamenti influissero sulla capacità della Shigella diinfettare il modello intestinale umano.
Una delle maggiori sfide nel condurre questo tipo di screening è il verificarsi di colli di bottiglia nella popolazione. Questi si verificano quando fattori come la carenza di nutrienti o la competizione per i siti di colonizzazione riducono temporaneamente il numero di batteri, indipendentemente dalla fitness dei singoli mutanti. Questo porta a una perdita stocastica di mutanti, che può distorcere i risultati dei grandi screening. Per compensare questa situazione, il team ha sviluppato un modello statistico che integra le informazioni di un gran numero di esperimenti su piccola scala per generare una mappa a livello di genoma. Questo ha permesso al team di generare la prima mappa completa dei geni che Shigella utilizza per invadere il tessuto intestinale umano.
Particolarmente sorprendente è stata la scoperta che alcuni cambiamenti negli acidi ribonucleici di trasferimento (tRNA) possono controllare l'attività del sistema di secrezione di tipo III, un complesso apparato che i batteri utilizzano per sprigionare le loro proprietà che causano malattie. La produzione di questi sistemi costa ai batteri molta energia, ed è per questo che li controllano in modo molto preciso. "In precedenza si sapeva che i batteri lo fanno, ad esempio, spegnendo il DNA estraneo o regolando il numero di alcuni plasmidi, piccole molecole circolari di DNA. Questo studio rivela ora un terzo meccanismo fondamentale con cui è possibile ottenere questo controllo", spiega Laura Jenniches, ricercatrice post-dottorato nel laboratorio di Barquist che ha condotto l'analisi statistica e computazionale.
"LaShigella possiede circa 5.000 geni, ma abbiamo scoperto che solo circa 100 di essi sono necessari al batterio per colonizzare i tessuti e causare un'infezione aggressiva. Questo elenco è una miniera d'oro per comprendere la progressione delle infezioni e per sviluppare nuovi trattamenti in grado di 'spegnere' il comportamento patogeno del batterio", afferma Mikael Sellin dell'Università di Uppsala, un altro degli autori senior dello studio. La tecnologia di screening utilizzata è trasferibile a molti altri agenti patogeni e consentirà in futuro studi realistici su tessuti umani. Lars Barquist guarda avanti: "Questo studio pone le basi tecniche necessarie per studiare un'ampia varietà di agenti patogeni in questi modelli organoidi realistici che catturano aspetti chiave della fisiologia umana in laboratorio".
Nota: questo articolo è stato tradotto utilizzando un sistema informatico senza intervento umano. LUMITOS offre queste traduzioni automatiche per presentare una gamma più ampia di notizie attuali. Poiché questo articolo è stato tradotto con traduzione automatica, è possibile che contenga errori di vocabolario, sintassi o grammatica. L'articolo originale in Inglese può essere trovato qui.
Pubblicazione originale
Di Martino ML, Jenniches L, Bhetwal A, Eriksson J, Lopes ACC, Ntokaki A, Pasqua M, Sundbom M, Skogar M, Graf W, Webb DL, Hellström PM, Mateus A, Barquist L, Sellin ME (2025): A scalable gut epithelial organoid model reveals the genome-wide colonization landscape of a human-adapted pathogen. Nature Genetics