Des mini-organes cultivés révèlent les armes des bactéries agressives
L'étude ouvre la voie à l'utilisation de mini-organes humains cultivés pour étudier un large éventail d'autres infections graves
Grâce à des intestins miniatures cultivés en laboratoire, des chercheurs de l'Institut Helmholtz de recherche sur les infections à ARN (HIRI) de Würzburg, en collaboration avec l'université d'Uppsala en Suède, ont réussi à cartographier la manière dont la bactérie agressive Shigella infecte l'intestin humain. L'étude, publiée dans la revue Nature Genetics, ouvre la voie à l'utilisation de mini-organes humains cultivés pour étudier un large éventail d'autres infections graves.
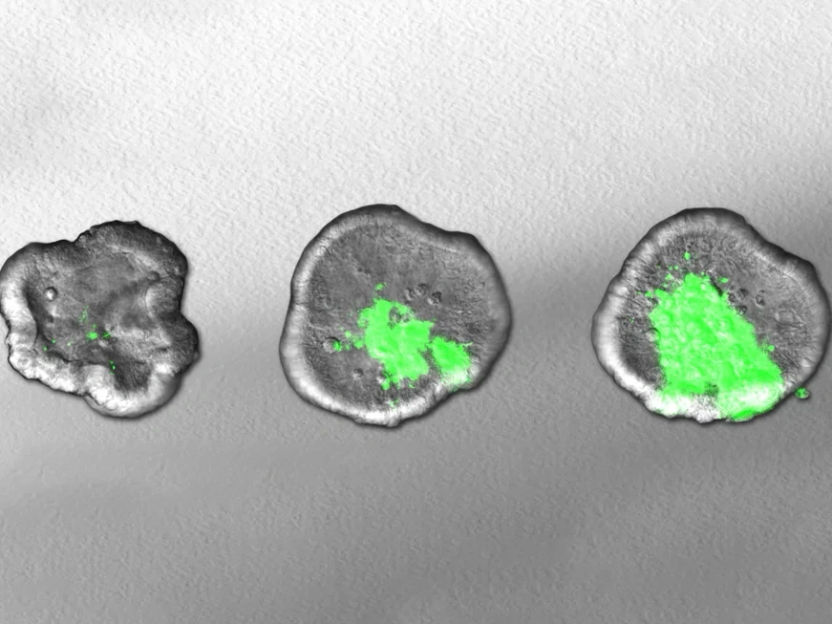
L'image montre comment la bactérie Shigella (en vert) se propage au fil du temps dans un intestin miniature cultivé en laboratoire.
© Maria Letizia Di Martino
Il est difficile de comprendre comment les bactéries spécifiques à l'homme nous rendent malades. Dans une nouvelle étude publiée dans la revue Nature Genetics, des chercheurs montrent qu'il est désormais possible d'utiliser des mini-organes cultivés, appelés organoïdes, pour cartographier la façon dont les bactéries colonisent la muqueuse intestinale humaine. L'équipe de l'Institut Helmholtz de recherche sur les infections à base d'ARN (HIRI) de Würzburg, un site du Centre Helmholtz de Braunschweig pour la recherche sur les infections (HZI), en coopération avec l'université Julius-Maximilians de Würzburg (JMU) et l'université d'Uppsala, s'est concentrée spécifiquement sur Shigella flexneri. Cette bactérie provoque de graves inflammations intestinales chez l'homme et est responsable de nombreux décès par an, en particulier chez les jeunes enfants.
"Nos résultats donnent une image plus réaliste des facteurs qui influencent la colonisation de Shigella dans nos intestins. Cela pourrait permettre de nouvelles approches thérapeutiques à l'avenir", déclare Lars Barquist, scientifique associé à HIRI et professeur à l'université de Toronto au Canada, et coauteur principal de l'étude. "Pour la première fois, nous avons pu cartographier les gènes dont Shigella a besoin pour provoquer une infection en utilisant un modèle humain qui imite le tissu intestinal. L'étude démontre également que les mini-organes humains cultivés peuvent désormais être utilisés pour étudier une variété d'infections graves, en particulier celles pour lesquelles le manque de modèles animaux de laboratoire a précédemment limité la recherche", ajoute Maria Letizia Di Martino de l'université d'Uppsala, qui a dirigé la mise au point du système expérimental.
Des modèles intestinaux dérivés de cellules souches
Les bactériesShigella sont des agents pathogènes invasifs qui s'attaquent aux tissus de l'organisme en utilisant diverses "armes" pour envahir la muqueuse intestinale et manipuler les fonctions du système immunitaire de l'organisme. Dans l'étude actuelle, les chercheurs se sont attachés à identifier les gènes responsables de la production de ces armes. Pour ce faire, ils ont généré des organoïdes intestinaux - des modèles intestinaux miniatures - cultivés à partir de cellules souches humaines purifiées à partir de déchets chirurgicaux. Ils ont ensuite utilisé une méthode qui élimine de manière aléatoire des gènes bactériens et ont testé comment ces changements affectaient la capacité de Shigellaà infecter le modèle intestinal humain.
L'un des plus grands défis de ce type de dépistage est l'apparition de goulets d'étranglement au niveau de la population. Ceux-ci surviennent lorsque des facteurs tels que la carence en nutriments ou la concurrence pour les sites de colonisation diminuent temporairement le nombre de bactéries, quelle que soit l'aptitude des mutants individuels. Il en résulte une perte stochastique de mutants, qui peut fausser les résultats des grands cribles. Pour compenser ce phénomène, l'équipe a mis au point un modèle statistique qui intègre les informations provenant d'un grand nombre d'expériences à petite échelle afin de générer une carte à l'échelle du génome. Cela a permis à l'équipe de dresser la première carte complète des gènes utilisés par Shigella pour envahir le tissu intestinal humain.
La découverte que certains changements dans les acides ribonucléiques de transfert (ARNt) peuvent contrôler l'activité du système de sécrétion de type III - un appareil complexe que les bactéries utilisent pour libérer leurs propriétés pathogènes - a été particulièrement surprenante. La production de ces systèmes coûte beaucoup d'énergie aux bactéries, c'est pourquoi elles les contrôlent très précisément. "On savait déjà que les bactéries y parvenaient, par exemple, en désactivant l'ADN étranger ou en régulant le nombre de certains plasmides, de petites molécules d'ADN circulaires. Cette étude révèle maintenant un troisième mécanisme fondamental par lequel ce contrôle peut être réalisé", explique Laura Jenniches, la chercheuse postdoctorale du laboratoire Barquist qui a dirigé l'analyse statistique et computationnelle.
"Shigella possède environ 5 000 gènes, mais nous avons découvert qu'une centaine d'entre eux seulement sont nécessaires pour que la bactérie colonise les tissus et provoque une infection agressive. Cette liste est une mine d'or pour comprendre la progression des infections et pour mettre au point de nouveaux traitements capables de "désactiver" le comportement pathogène de la bactérie", explique Mikael Sellin, de l'université d'Uppsala, un autre des auteurs principaux de l'étude. La technologie de dépistage utilisée est transférable à de nombreux autres agents pathogènes et permettra à l'avenir de réaliser des études réalistes sur des tissus humains. Lars Barquist se projette dans l'avenir : "Cette étude jette les bases techniques nécessaires à l'étude d'une grande variété d'agents pathogènes dans ces modèles organoïdes réalistes qui capturent des aspects clés de la physiologie humaine en laboratoire."
Note: Cet article a été traduit à l'aide d'un système informatique sans intervention humaine. LUMITOS propose ces traductions automatiques pour présenter un plus large éventail d'actualités. Comme cet article a été traduit avec traduction automatique, il est possible qu'il contienne des erreurs de vocabulaire, de syntaxe ou de grammaire. L'article original dans Anglais peut être trouvé ici.
Publication originale
Di Martino ML, Jenniches L, Bhetwal A, Eriksson J, Lopes ACC, Ntokaki A, Pasqua M, Sundbom M, Skogar M, Graf W, Webb DL, Hellström PM, Mateus A, Barquist L, Sellin ME (2025): A scalable gut epithelial organoid model reveals the genome-wide colonization landscape of a human-adapted pathogen. Nature Genetics