Miniórganos cultivados revelan las armas de las bacterias agresivas
El estudio abre la puerta al uso de miniórganos humanos cultivados para investigar una amplia gama de otras infecciones graves
Gracias a intestinos en miniatura cultivados en laboratorio, investigadores del Instituto Helmholtz para la Investigación de Infecciones Basadas en el ARN (HIRI) de Würzburg, en colaboración con la Universidad de Uppsala (Suecia), han logrado cartografiar cómo la agresiva bacteria Shigella infecta el intestino humano. El estudio, publicado en la revista Nature Genetics, abre la puerta al uso de miniórganos humanos cultivados para investigar una amplia gama de otras infecciones graves.
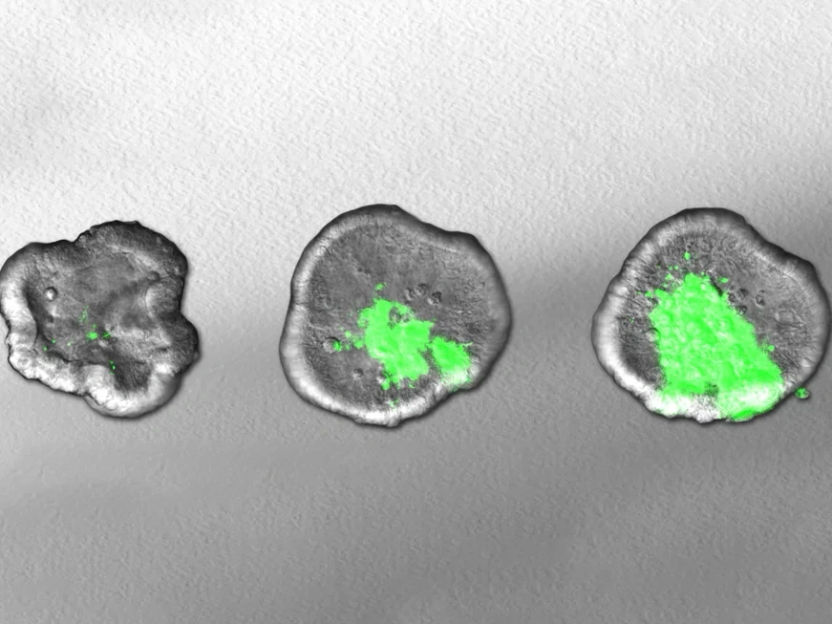
La imagen muestra cómo la bacteria Shigella (verde) se propaga con el tiempo en un intestino en miniatura cultivado en laboratorio.
© Maria Letizia Di Martino
Entender cómo nos enferman las bacterias específicas de los humanos es todo un reto. En un nuevo estudio publicado en Nature Genetics, los investigadores demuestran que ahora es posible utilizar miniórganos cultivados, los llamados organoides, para cartografiar cómo colonizan las bacterias la mucosa intestinal humana. El equipo del Instituto Helmholtz para la Investigación de Infecciones Basadas en el ARN (HIRI) de Würzburg, dependiente del Centro Helmholtz de Braunschweig para la Investigación de Infecciones (HZI) en cooperación con la Julius-Maximilians-Universität Würzburg (JMU), y de la Universidad de Uppsala, se centró específicamente en la Shigella flexneri. Esta bacteria provoca una inflamación intestinal grave en los seres humanos y es responsable de muchas muertes al año, sobre todo entre los niños pequeños.
"Nuestros resultados proporcionan una imagen más realista de los factores que influyen en la colonización de Shigella en nuestros intestinos. Esto podría permitir nuevos enfoques terapéuticos en el futuro", afirma Lars Barquist, científico asociado del HIRI y profesor de la Universidad de Toronto (Canadá), y coautor principal del estudio. "Por primera vez, hemos podido cartografiar los genes que Shigella necesita para causar infección utilizando un modelo humano que imita el tejido intestinal". El estudio también demuestra que los mini-órganos humanos cultivados pueden utilizarse ahora para investigar una variedad de infecciones graves, en particular aquellas para las que la falta de modelos animales de laboratorio ha limitado previamente la investigación", añade Maria Letizia Di Martino, de la Universidad de Uppsala, que dirigió el desarrollo del sistema experimental.
Modelos intestinales derivados de células madre
La bacteriaShigella es un patógeno invasor que ataca los tejidos del organismo utilizando diversas "armas" para invadir la mucosa intestinal y manipular las funciones del sistema inmunitario del cuerpo. En el estudio actual, los investigadores se centraron en identificar los genes responsables de producir estas armas. Para ello, generaron organoides intestinales -modelos intestinales en miniatura- cultivados a partir de células madre humanas purificadas de material de desecho quirúrgico. A continuación utilizaron un método que elimina genes bacterianos al azar y comprobaron cómo afectaban estos cambios a la capacidad de Shigellapara infectar el modelo intestinal humano.
Uno de los mayores retos a la hora de realizar este tipo de cribado es la aparición de cuellos de botella en la población. Estos surgen cuando factores como la deficiencia de nutrientes o la competencia por los lugares de colonización disminuyen temporalmente el número de bacterias, independientemente de la aptitud de los mutantes individuales. Esto conduce a una pérdida estocástica de mutantes, que puede distorsionar los resultados de grandes cribas. Para compensarlo, el equipo ha desarrollado un modelo estadístico que integra la información de un gran número de experimentos a pequeña escala para generar un mapa de todo el genoma. Esto permitió al equipo generar el primer mapa completo de los genes que la Shigella utiliza para invadir el tejido intestinal humano.
Especialmente sorprendente fue el descubrimiento de que ciertos cambios en los ácidos ribonucleicos de transferencia (ARNt) pueden controlar la actividad del sistema de secreción de tipo III, un complejo aparato que las bacterias utilizan para dar rienda suelta a sus propiedades patógenas. La producción de estos sistemas cuesta mucha energía a las bacterias, por lo que los controlan con gran precisión. "Hasta ahora se sabía que las bacterias lo hacen, por ejemplo, desactivando el ADN extraño o regulando el número de ciertos plásmidos, pequeñas moléculas circulares de ADN. Este estudio revela ahora un tercer mecanismo fundamental por el que se puede lograr este control", explica Laura Jenniches, la investigadora postdoctoral del laboratorio de Barquist que dirigió el análisis estadístico y computacional.
"Shigella tiene unos 5.000 genes, pero descubrimos que sólo unos 100 de ellos son necesarios para que la bacteria colonice tejidos y cause una infección agresiva. Esta lista es una mina de oro para comprender la progresión de las infecciones y desarrollar nuevos tratamientos que puedan 'desactivar' el comportamiento patógeno de la bacteria", afirma Mikael Sellin, de la Universidad de Uppsala, otro de los autores principales del estudio. La tecnología de cribado utilizada es transferible a muchos otros patógenos y permitirá en el futuro realizar estudios realistas en tejidos humanos". Lars Barquist mira hacia el futuro: "Este estudio sienta las bases técnicas necesarias para investigar una amplia variedad de patógenos en estos modelos organoides realistas que capturan aspectos clave de la fisiología humana en el laboratorio."
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.
Publicación original
Di Martino ML, Jenniches L, Bhetwal A, Eriksson J, Lopes ACC, Ntokaki A, Pasqua M, Sundbom M, Skogar M, Graf W, Webb DL, Hellström PM, Mateus A, Barquist L, Sellin ME (2025): A scalable gut epithelial organoid model reveals the genome-wide colonization landscape of a human-adapted pathogen. Nature Genetics