Os mini-órgãos cultivados revelam as armas das bactérias agressivas
O estudo abre a porta à utilização de mini-órgãos humanos em cultura para investigar uma vasta gama de outras infecções graves
Anúncios
Graças a intestinos em miniatura cultivados em laboratório, os investigadores do Instituto Helmholtz para a Investigação de Infecções Baseadas em ARN (HIRI), em Würzburg, em colaboração com a Universidade de Uppsala, na Suécia, conseguiram mapear a forma como a agressiva bactéria Shigella infecta o intestino humano. O estudo, publicado na revista Nature Genetics, abre a porta à utilização de mini-órgãos humanos em cultura para investigar uma vasta gama de outras infecções graves.
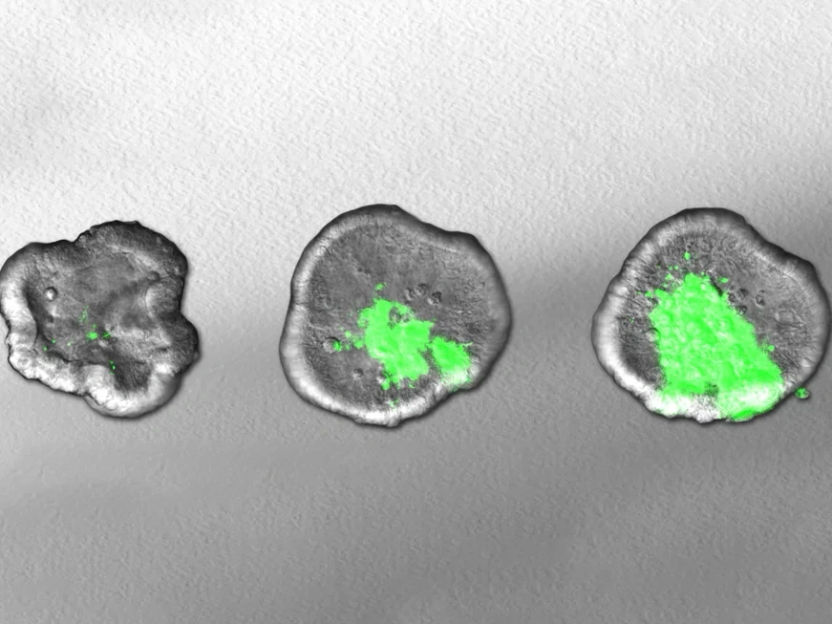
A imagem mostra como a bactéria Shigella (verde) se espalha ao longo do tempo num intestino em miniatura cultivado em laboratório.
© Maria Letizia Di Martino
Compreender como é que as bactérias específicas do ser humano nos fazem adoecer é um desafio. Num novo estudo publicado na revista Nature Genetics, os investigadores mostram que é agora possível utilizar mini-órgãos cultivados, os chamados organóides, para mapear a forma como as bactérias colonizam a mucosa intestinal humana. A equipa do Instituto Helmholtz para a Investigação de Infecções com base em ARN (HIRI) em Würzburg, uma unidade do Centro Helmholtz de Braunschweig para a Investigação de Infecções (HZI), em cooperação com a Julius-Maximilians-Universität Würzburg (JMU) e a Universidade de Uppsala, concentrou-se especificamente na Shigella flexneri. Esta bactéria provoca uma inflamação intestinal grave nos seres humanos e é responsável por muitas mortes por ano, sobretudo entre as crianças.
"Os nossos resultados fornecem uma imagem mais realista dos factores que influenciam a colonização da Shigella nos nossos intestinos. Isto poderá permitir novas abordagens de tratamento no futuro", afirma Lars Barquist, cientista associado do HIRI e professor na Universidade de Toronto, no Canadá, e coautor principal do estudo. "Pela primeira vez, conseguimos mapear os genes de que a Shigella necessita para causar a infeção, utilizando um modelo humano que imita o tecido intestinal. O estudo demonstra também que os mini-órgãos humanos cultivados podem agora ser utilizados para investigar uma série de infecções graves, em especial aquelas para as quais a falta de modelos animais de laboratório limitava anteriormente a investigação", acrescenta Maria Letizia Di Martino, da Universidade de Uppsala, que liderou o desenvolvimento do sistema experimental.
Modelos intestinais derivados de células estaminais
As bactériasShigella são agentes patogénicos invasivos que atacam os tecidos do corpo utilizando uma variedade de "armas" para invadir a mucosa intestinal e manipular as funções do sistema imunitário do corpo. No estudo atual, os investigadores concentraram-se em identificar os genes responsáveis pela produção destas armas. Para isso, geraram organóides intestinais - modelos intestinais em miniatura - cultivados a partir de células estaminais humanas purificadas de resíduos cirúrgicos. Em seguida, utilizaram um método que elimina aleatoriamente os genes bacterianos e testaram a forma como essas alterações afectavam a capacidade da Shigellade infetar o modelo intestinal humano.
Um dos maiores desafios na realização deste tipo de rastreio é a ocorrência de estrangulamentos populacionais. Estes surgem quando factores como a deficiência de nutrientes ou a competição por locais de colonização diminuem temporariamente o número de bactérias, independentemente da aptidão dos mutantes individuais. Isto leva a uma perda estocástica de mutantes, que pode distorcer os resultados de grandes rastreios. Para compensar esta situação, a equipa desenvolveu um modelo estatístico que integra a informação de um grande número de experiências de pequena escala para gerar um mapa de todo o genoma. Isto permitiu à equipa gerar o primeiro mapa completo dos genes que a Shigella utiliza para invadir o tecido intestinal humano.
Particularmente surpreendente foi a descoberta de que certas alterações nos ácidos ribonucleicos de transferência (ARNt) podem controlar a atividade do sistema de secreção do tipo III - um aparelho complexo que as bactérias utilizam para libertar as suas propriedades causadoras de doenças. Estes sistemas custam às bactérias uma grande quantidade de energia para serem produzidos, razão pela qual as bactérias os controlam com grande precisão. "Sabia-se anteriormente que as bactérias o fazem, por exemplo, desligando o ADN estranho ou regulando o número de certos plasmídeos, pequenas moléculas circulares de ADN. Este estudo revela agora um terceiro mecanismo fundamental que permite este controlo", explica Laura Jenniches, investigadora de pós-doutoramento do laboratório de Barquist, responsável pela análise estatística e computacional.
"A Shigella tem cerca de 5.000 genes, mas descobrimos que apenas cerca de 100 deles são necessários para a bactéria colonizar os tecidos e causar uma infeção agressiva. Esta lista é uma mina de ouro para compreender a progressão das infecções e para desenvolver novos tratamentos que possam 'desligar' o comportamento patogénico da bactéria", diz Mikael Sellin da Universidade de Uppsala, outro dos autores principais do estudo. A tecnologia de rastreio utilizada é transferível para muitos outros agentes patogénicos e permitirá, no futuro, a realização de estudos realistas em tecidos humanos. Lars Barquist olha para o futuro: "Este estudo estabelece as bases técnicas necessárias para investigar uma grande variedade de agentes patogénicos nestes modelos realistas de organóides que captam aspectos fundamentais da fisiologia humana no laboratório".
Observação: Este artigo foi traduzido usando um sistema de computador sem intervenção humana. A LUMITOS oferece essas traduções automáticas para apresentar uma gama mais ampla de notícias atuais. Como este artigo foi traduzido com tradução automática, é possível que contenha erros de vocabulário, sintaxe ou gramática. O artigo original em Inglês pode ser encontrado aqui.
Publicação original
Di Martino ML, Jenniches L, Bhetwal A, Eriksson J, Lopes ACC, Ntokaki A, Pasqua M, Sundbom M, Skogar M, Graf W, Webb DL, Hellström PM, Mateus A, Barquist L, Sellin ME (2025): A scalable gut epithelial organoid model reveals the genome-wide colonization landscape of a human-adapted pathogen. Nature Genetics