Analyse d'images microscopiques : un nouveau logiciel libre rend les modèles d'IA plus légers et plus écologiques
Aucune connaissance particulière n'est requise
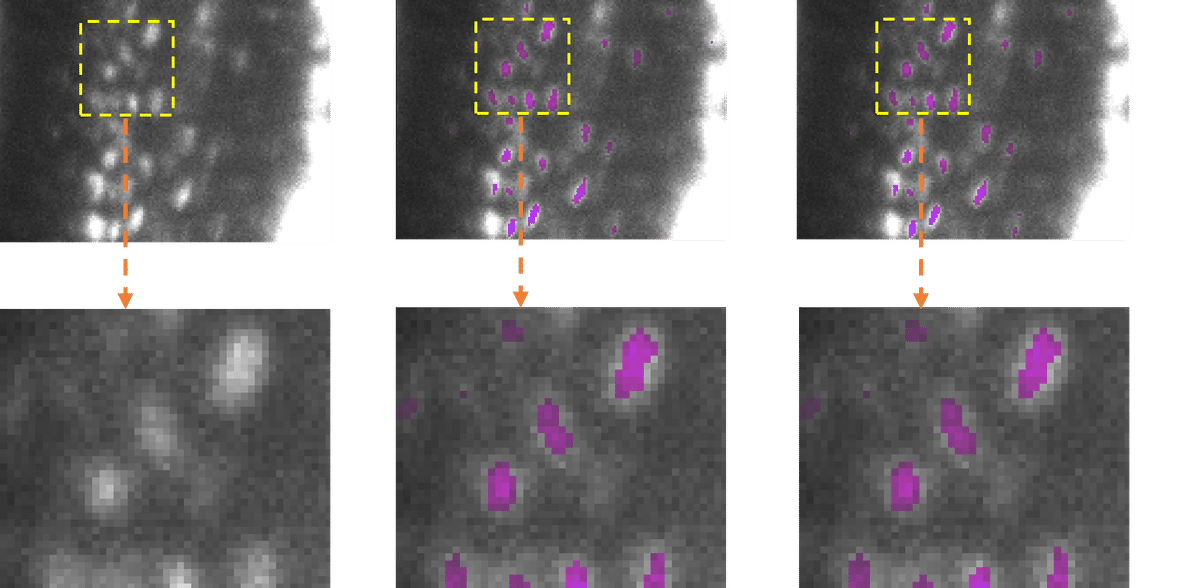
L'intelligence artificielle (IA) est devenue une composante indispensable de l'analyse des données microscopiques. Cependant, alors que les modèles d'IA s'améliorent et deviennent plus complexes, la puissance de calcul et la consommation d'énergie associée augmentent également. Des chercheurs du Leibniz-Institut für Analytische Wissenschaften (ISAS) et de l'Université de Pékin ont donc créé un logiciel de compression gratuit qui permet aux scientifiques d'exécuter les modèles d'IA de bio-imagerie existants plus rapidement et en consommant beaucoup moins d'énergie. Les chercheurs viennent de présenter leur boîte à outils conviviale appelée EfficientBioAI (open source) dans Nature Methods.
Les techniques modernes de microscopie produisent un grand nombre d'images à haute résolution, et les ensembles de données individuels peuvent en comprendre des milliers. Les scientifiques utilisent souvent des logiciels assistés par l'IA pour analyser de manière fiable ces ensembles de données. Cependant, à mesure que les modèles d'IA deviennent plus complexes, la latence (temps de traitement) des images peut augmenter de manière significative. "Une latence élevée du réseau, par exemple avec des images particulièrement grandes, entraîne une augmentation de la puissance de calcul et, en fin de compte, de la consommation d'énergie", explique le Dr Jianxu Chen, responsable du groupe de recherche junior AMBIOM - Analysis of Microscopic BIOMedical Images (Analyse d'images microscopiques biomédicales) à l'ISAS.
Une technique bien connue trouve de nouvelles applications
Pour éviter les temps de latence élevés dans l'analyse d'images, en particulier sur les appareils à puissance de calcul limitée, les chercheurs utilisent des algorithmes sophistiqués pour compresser les modèles d'intelligence artificielle. Cela signifie qu'ils réduisent la quantité de calculs dans les modèles tout en conservant une précision de prédiction comparable. "La compression des modèles est une technique largement utilisée dans le domaine du traitement numérique de l'image, connu sous le nom de vision par ordinateur, et de l'IA pour rendre les modèles plus légers et plus écologiques", explique Chen. Les chercheurs combinent diverses stratégies pour réduire la consommation de mémoire, accélérer l'inférence du modèle, le "processus de pensée" du modèle, et ainsi économiser de l'énergie. L'élagage, par exemple, est utilisé pour supprimer les nœuds excédentaires du réseau neuronal. "Ces techniques sont souvent encore inconnues de la communauté de la bio-imagerie. C'est pourquoi nous avons voulu développer une solution simple et prête à l'emploi pour les appliquer aux outils d'IA courants en bioimagerie", explique Yu Zhou, premier auteur de l'article et étudiant en doctorat à AMBIOM.
Des économies d'énergie pouvant atteindre 81 % environ
Pour mettre leur nouvelle boîte à outils à l'épreuve, les chercheurs dirigés par Chen ont testé leur logiciel sur plusieurs applications réelles. Avec différents matériels et différentes tâches d'analyse de bio-imagerie, les techniques de compression ont permis de réduire significativement la latence et la consommation d'énergie de 12,5 à 80,6 %. "Nos tests montrent qu'EfficientBioAI peut augmenter de manière significative l'efficacité des réseaux neuronaux en bio-imagerie sans limiter la précision des modèles", résume Chen. Il illustre les économies d'énergie en prenant pour exemple le modèle CellPose couramment utilisé : si un millier d'utilisateurs se servaient de la boîte à outils pour compresser le modèle et l'appliquer à l'ensemble de données Jump Target ORF (environ un million d'images de cellules prises au microscope), ils pourraient économiser l'énergie équivalente aux émissions d'un voyage en voiture d'environ 7 300 miles (approximativement 11 750 kilomètres).
Aucune connaissance particulière n'est requise
Les auteurs souhaitent rendre EfficientBioAI accessible au plus grand nombre possible de scientifiques travaillant dans le domaine de la recherche biomédicale. Les chercheurs peuvent installer le logiciel et l'intégrer de manière transparente dans les bibliothèques PyTorch existantes (bibliothèque de programmes open-source pour le langage de programmation Python). Pour certains modèles largement utilisés, comme Cellpose, les chercheurs peuvent donc utiliser le logiciel sans avoir à modifier eux-mêmes le code. Pour répondre aux demandes de modifications spécifiques, le groupe fournit également plusieurs démonstrations et tutoriels. Avec seulement quelques lignes de code modifiées, la boîte à outils peut également être appliquée à des modèles d'IA personnalisés.
Note: Cet article a été traduit à l'aide d'un système informatique sans intervention humaine. LUMITOS propose ces traductions automatiques pour présenter un plus large éventail d'actualités. Comme cet article a été traduit avec traduction automatique, il est possible qu'il contienne des erreurs de vocabulaire, de syntaxe ou de grammaire. L'article original dans Anglais peut être trouvé ici.