Análisis de imágenes microscópicas: un nuevo software de código abierto hace que los modelos de IA sean más ligeros y ecológicos
No se requieren conocimientos especiales
La inteligencia artificial (IA) se ha convertido en un componente indispensable en el análisis de datos microscópicos. Sin embargo, aunque los modelos de IA son cada vez mejores y más complejos, la potencia de cálculo y el consumo de energía asociado también aumentan. Por ello, investigadores del Leibniz-Institut für Analytische Wissenschaften (ISAS) y la Universidad de Pekín han creado un software de compresión gratuito que permite a los científicos ejecutar los modelos de IA de bioimagen existentes más rápidamente y con un consumo de energía significativamente menor. Los investigadores han presentado ahora en Nature Methods su caja de herramientas de fácil manejo, denominada EfficientBioAI (de código abierto).
Las técnicas modernas de microscopía producen un gran número de imágenes de alta resolución, y los conjuntos de datos individuales pueden constar de miles de ellas. Los científicos suelen utilizar programas informáticos basados en IA para analizar de forma fiable estos conjuntos de datos. Sin embargo, a medida que los modelos de IA se hacen más complejos, la latencia (tiempo de procesamiento) de las imágenes puede aumentar considerablemente. "Una latencia de red elevada, por ejemplo con imágenes especialmente grandes, conlleva una mayor potencia de cálculo y, en última instancia, un mayor consumo de energía", afirma el Dr. Jianxu Chen, responsable del grupo de investigación AMBIOM - Analysis of Microscopic BIOMedical Images junior en ISAS.
Una técnica bien conocida encuentra nuevas aplicaciones
Para evitar la alta latencia en el análisis de imágenes, sobre todo en dispositivos con potencia de cálculo limitada, los investigadores utilizan sofisticados algoritmos para comprimir los modelos de IA. Esto significa que reducen la cantidad de cálculos de los modelos sin perder una precisión de predicción comparable. "La compresión de modelos es una técnica muy utilizada en el campo del procesamiento digital de imágenes, conocido como visión por ordenador, y la IA para hacer los modelos más ligeros y ecológicos", explica Chen. Los investigadores combinan varias estrategias para reducir el consumo de memoria, acelerar la inferencia del modelo -el "proceso de pensamiento" del modelo- y ahorrar así energía. La poda, por ejemplo, se utiliza para eliminar los nodos sobrantes de la red neuronal. "Estas técnicas suelen ser aún desconocidas en la comunidad de la bioimagen. Por eso queríamos desarrollar una solución sencilla y lista para usar que pudiera aplicarse a las herramientas de IA más comunes en bioimagen", explica Yu Zhou, primer autor del artículo y estudiante de doctorado en AMBIOM.
Ahorro de energía de hasta un 81
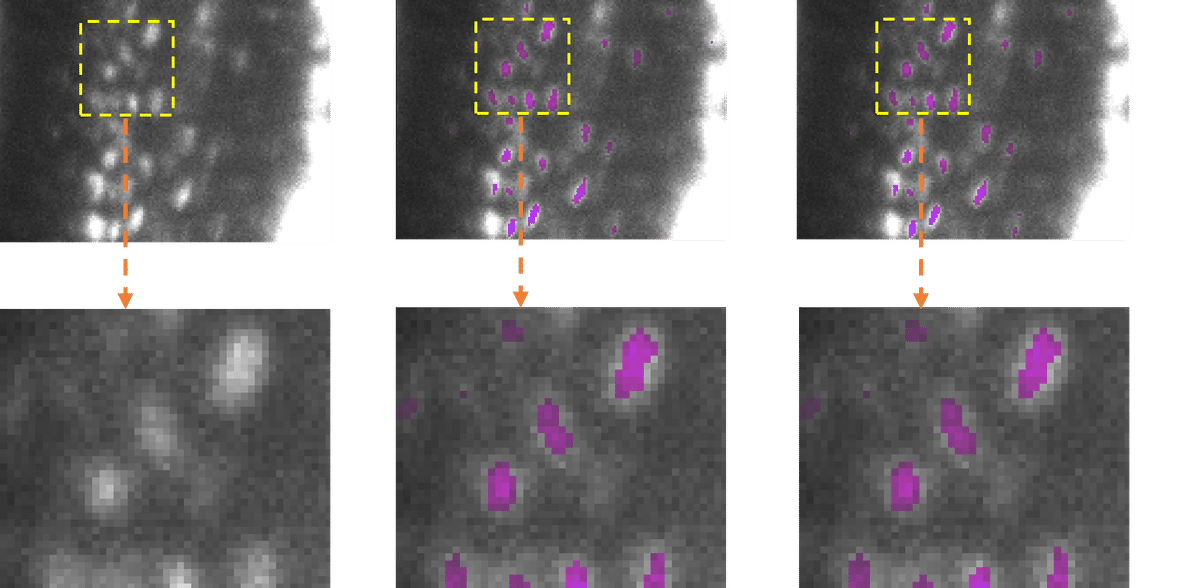
Para poner a prueba su nuevo conjunto de herramientas, los investigadores dirigidos por Chen probaron su software en varias aplicaciones de la vida real. Con hardware diferente y diversas tareas de análisis de bioimágenes, las técnicas de compresión consiguieron reducir significativamente la latencia y recortar el consumo de energía entre un 12,5 y un 80,6 por ciento. "Nuestras pruebas demuestran que EfficientBioAI puede aumentar significativamente la eficiencia de las redes neuronales en bioimagen sin limitar la precisión de los modelos", resume Chen. Chen ilustra el ahorro de energía utilizando como ejemplo el modelo CellPose, de uso común: si mil usuarios utilizaran la caja de herramientas para comprimir el modelo y aplicarlo al conjunto de datos Jump Target ORF (alrededor de un millón de imágenes de células obtenidas con microscopio), podrían ahorrar una energía equivalente a las emisiones de un viaje en coche de unas 7.300 millas (unos 11.750 kilómetros).
No se necesitan conocimientos especiales
Los autores desean que EfficientBioAI sea accesible al mayor número posible de científicos dedicados a la investigación biomédica. Los investigadores pueden instalar el software e integrarlo sin problemas en las bibliotecas PyTorch existentes (biblioteca de programas de código abierto para el lenguaje de programación Python). Para algunos modelos ampliamente utilizados, como Cellpose, los investigadores pueden por tanto utilizar el software sin tener que realizar ellos mismos ningún cambio en el código. Para apoyar las solicitudes de cambios específicos, el grupo también proporciona varias demostraciones y tutoriales. Con unas pocas líneas de código modificadas, la caja de herramientas puede aplicarse también a modelos de IA personalizados.
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.