Un nouveau logiciel basé sur l'IA permet une imagerie rapide et fiable des protéines dans les cellules
Des chercheurs ont programmé un outil qui reconnaît et sélectionne avec précision les protéines dans la cryo-tomographie électronique, remplaçant ainsi la sélection manuelle fastidieuse.
La cryo-tomographie électronique (cryo-ET) apparaît comme une technique puissante pour fournir des images 3D détaillées des environnements cellulaires et des biomolécules qui y sont enfermées. Cependant, l'un des défis de cette méthodologie est l'identification des molécules de protéines dans les images en vue d'un traitement ultérieur. Une équipe de recherche autour de Stefan Raunser, directeur du MPI de physiologie moléculaire de Dortmund, dirigée par Thorsten Wagner, a mis au point un logiciel permettant de repérer les protéines dans les volumes cellulaires encombrés. Le nouvel outil open-source, appelé TomoTwin, est basé sur l'apprentissage métrique profond et permet aux scientifiques de localiser plusieurs protéines avec une grande précision et un débit élevé sans avoir à créer ou à réapprendre manuellement le réseau à chaque fois.

Image symbolique
Computer-generated image
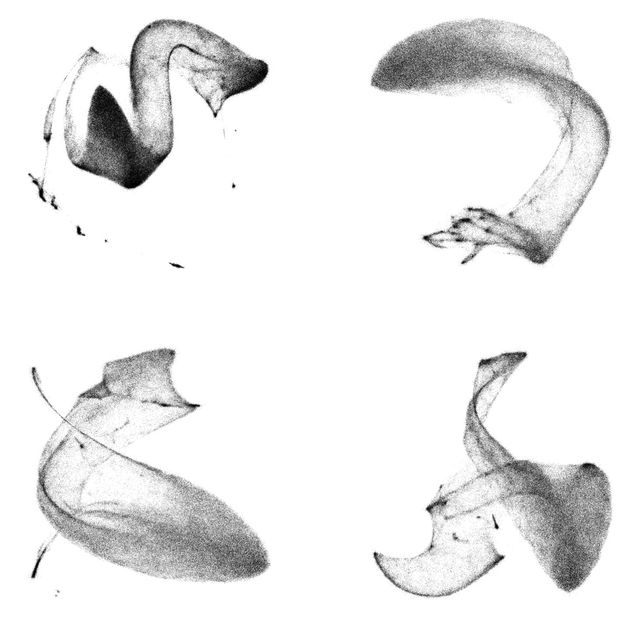
Carte de traitement TomoTwin pour un tomogramme aplati en 2D. Les particules de différentes macromolécules sont disposées dans la carte en fonction de leur structure, ce qui permet aux utilisateurs d'identifier et de localiser les différentes macromolécules à l'intérieur des cellules.
MPI of Molecular Physiology

Les premiers auteurs Gavin Rice (à gauche) et Thorsten Wagner (à droite).
MPI of Molecular Physiology


Plus il y en a, mieux c'est
"TomoTwin ouvre la voie à l'identification et à la localisation automatisées des protéines directement dans leur environnement cellulaire, élargissant ainsi le potentiel de la cryo-ET", explique Gavin Rice, coauteur de la publication. La cryo-ET a le potentiel de déchiffrer le fonctionnement des biomolécules au sein d'une cellule et, par conséquent, de dévoiler les fondements de la vie et l'origine des maladies.
Dans une expérience de cryo-ET, les scientifiques utilisent un microscope électronique à transmission pour obtenir des images en 3D, appelées tomogrammes, du volume cellulaire contenant des biomolécules complexes. Pour obtenir une image plus détaillée de chaque protéine, ils font la moyenne d'autant de copies que possible - un peu comme les photographes qui prennent la même photo à des expositions différentes pour les combiner ensuite dans une image parfaitement exposée. Il est essentiel d'identifier et de localiser correctement les différentes protéines dans l'image avant d'en faire la moyenne. "Les scientifiques peuvent obtenir des centaines de tomogrammes par jour, mais nous manquions d'outils pour identifier complètement les molécules qu'ils contiennent", explique M. Rice.
Sélection manuelle
Jusqu'à présent, les chercheurs ont utilisé des algorithmes basés sur des modèles de structures moléculaires déjà connues pour rechercher des correspondances dans les tomogrammes, mais ces algorithmes ont tendance à être entachés d'erreurs. L'identification manuelle des molécules est une autre option qui garantit une sélection de haute qualité, mais qui prend des jours, voire des semaines, par ensemble de données.
Une autre possibilité serait d'utiliser une forme d'apprentissage automatique supervisé. Ces outils peuvent être très précis mais manquent actuellement de convivialité, car ils nécessitent l'étiquetage manuel de milliers d'exemples pour former le logiciel à chaque nouvelle protéine, une tâche presque impossible pour les petites molécules biologiques dans un environnement cellulaire encombré.
TomoTwin
Le nouveau logiciel TomoTwin permet de surmonter bon nombre de ces obstacles : Il apprend à sélectionner les molécules de forme similaire dans un tomogramme et les cartographie dans un espace géométrique - le système est récompensé lorsqu'il place des protéines similaires à proximité les unes des autres et pénalisé dans le cas contraire. Dans la nouvelle carte, les chercheurs peuvent isoler et identifier avec précision les différentes protéines et les utiliser pour les localiser à l'intérieur de la cellule. "L'un des avantages de TomoTwin est que nous fournissons un modèle de prélèvement pré-entraîné", explique M. Rice. En supprimant l'étape de formation, le logiciel peut même fonctionner sur des ordinateurs locaux - alors que le traitement d'un tomogramme nécessite habituellement 60 à 90 minutes, le temps d'exécution sur le superordinateur MPI Raven est réduit à 15 minutes par tomogramme.
TomoTwin permet aux chercheurs de sélectionner des dizaines de tomogrammes dans le temps qu'il faut pour en sélectionner un seul manuellement, augmentant ainsi le débit de données et le taux de moyennage pour obtenir une meilleure image. Le logiciel peut actuellement localiser les protéines globulaires ou les complexes protéiques de plus de 150 kilodaltons dans les cellules ; à l'avenir, le groupe de Raunser souhaite inclure les protéines membranaires, les protéines filamenteuses et les protéines de plus petite taille.
Note: Cet article a été traduit à l'aide d'un système informatique sans intervention humaine. LUMITOS propose ces traductions automatiques pour présenter un plus large éventail d'actualités. Comme cet article a été traduit avec traduction automatique, il est possible qu'il contienne des erreurs de vocabulaire, de syntaxe ou de grammaire. L'article original dans Anglais peut être trouvé ici.