Zwei Tage Messbetrieb für die Suche nach dem richtigen Schlüssel
BESSY II startet neue Messkampagne, um die Suche nach Corona-Wirkstoffen zu beschleunigen
Anzeigen
Die Berliner Synchrotronquelle BESSY II des Helmholtz-Zentrum Berlin (HZB) nimmt für zwei Tage den Betrieb wieder auf. Mit der intensiven Röntgenstrahlung von BESSY II wollen Forscher nach Wirkstoffen gegen das Coronavirus SARS-CoV2 suchen. Fast zweihundert Proben aus einem wichtigen Protein des Virus werden in den kommenden Stunden untersucht. Die Proben sind mit unterschiedlichen Molekülen getränkt, die als Bestandteile von Wirkstoffen in Frage kommen. Die Analysen werden zeigen, ob bestimmte Moleküle besonders gut an das Proteinmolekül andocken und damit die Vermehrung des Virus behindern können. Diese Moleküle könnten Bestandteile eines künftigen Wirkstoffs werden.
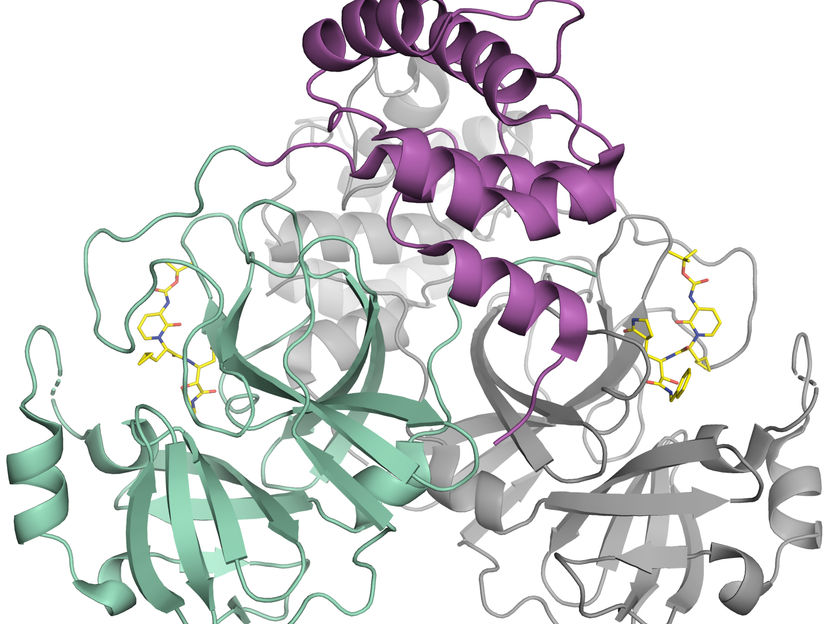
Schematische Darstellung der Coronavirus-Protease. Das Enzym kommt als Dimer bestehend aus zwei identischen Molekülen vor. Ein Teil des Dimers ist in Farbe dargestellt (grün und violett), der andere in grau. Das kleine Molekül in gelb bindet an das aktive Zentrum der Protease und könnte als Blaupause für einen Hemmstoff dienen.
© Helena Tabermann/HZB
Bereits im Februar hatten Prof. Dr. Rolf Hilgenfeld (Universität Lübeck) und sein Team an der Röntgenquelle BESSY II die Struktur eines Proteins des SARS-CoV-2-Virus entschlüsselt. Es handelt sich dabei um die Hauptprotease, die an der Vermehrung der Viren beteiligt ist. Für die Funktion dieser Protease ist ihre dreidimensionale Gestalt entscheidend, insbesondere die Umgebung von so genannten aktiven Zentren im Molekül.
3D-Struktur der Protease an BESSY II entschlüsselt
Durch Messungen an den MX-Messplätzen an BESSY II konnte die 3D-Struktur der Protease im Detail ermittelt werden. „Das war ein großer Durchbruch“, sagt Dr. Manfred Weiss, der das MX-Team an BESSY II leitet. „Sobald die 3D-Struktur bekannt ist, kann man gezielt nach Wirkstoffen suchen, die diese Protease blockieren und damit die Vermehrung der Viren verhindern“, erklärt Weiss.
Systematische Suche nach Wirkstoffen
An BESSY II hat das MX-Team seit einigen Jahren ein Verfahren aufgebaut, das nun eine systematische Suche nach Wirkstoffen ermöglicht: Das Fragment-Screening. Denn ein geeigneter Wirkstoff muss Bestandteile oder Fragmente besitzen, die genau auf die 3D-Struktur des Proteins zugeschnitten sind. Sie müssen wie ein Schlüssel ins Schloss passen, damit sie die Funktion des Proteins behindern.
Eine Fragment-Bibliothek besteht aus hunderten von Molekülgruppen, die als Bestandteile von Wirkstoffen in Frage kommen. Und wie ein Einbrecher einen Dietrich nutzt, um nach und nach herauszufinden, wie der perfekte Schlüssel für den Safe aussieht, so kann die Forschung das Fragment-Screening nutzen. Nach und nach werden damit unterschiedliche Moleküle getestet, bis die besten Komponenten für einen passenden Wirkstoff identifziert sind.
Fast 200 Proben werden nun analysiert
Dr. Linlin Zhang aus dem Lübecker Team hat nun eine große Anzahl von Proteinkristallen hergestellt und jede Probe mit einer anderen Verbindung aus der Fragment-Bibliothek getränkt. Diese Proben werden nun an BESSY II untersucht. Aus den Ergebnissen lässt sich ermitteln, welche Fragmente überhaupt im aktiven Zentrum der viralen Protease andocken. Diese Fragmente kommen dann als Bestandteile für einen Wirkstoff in Frage.
„Natürlich müssen im Anschluss weitere Experimente und Messreihen erfolgen, sowohl Versuche am Tiermodell als auch an menschlichen Zellkulturen“, sagt Weiss. Aber angesichts der unendlichen Anzahl von chemischen Verbindungen bietet diese systematische Vorauswahl an vielversprechenden Bausteinen die Chance auf eine enorme Beschleunigung.
„Wir haben zunächst mit der kleineren Bibliothek aus 96 Fragmenten begonnen“, sagt Weiss. „Aber sollte es nicht genug Treffer geben, können wir auch auf eine deutlich größere Bibliothek zugreifen“.