L'analyse scientifique des images pour tous
Un nouveau logiciel permet l'analyse automatisée des données d'image sans connaissances en programmation
Le logiciel JIPipe a été développé par des scientifiques de l'Institut Leibniz pour la recherche sur les produits naturels et la biologie des infections (Leibniz-HKI) et simplifie considérablement l'analyse des images générées par la recherche. Les utilisateurs peuvent créer des organigrammes en fonction des besoins de leur application et ainsi effectuer des analyses d'images automatiques en utilisant l'intelligence artificielle sans aucune connaissance en programmation. JIPipe est basé sur ImageJ, un programme standard d'analyse scientifique d'images microscopiques biomédicales. Les auteurs présentent maintenant leur développement dans Nature Methods.
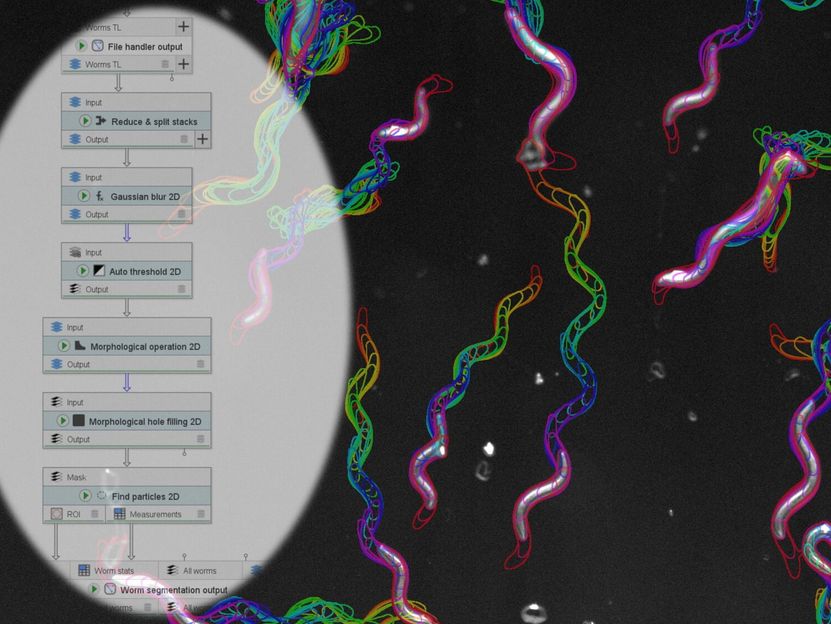
JIPipe peut être utilisé, par exemple, pour analyser les profils de déplacement des nématodes.
Hannah Büttner, Zoltán Cseresnyés, Ruman Gerst
Capture d'écran d'une analyse JIPipe de la survie des nématodes.
Zoltán Cseresnyés/Leibniz-HKI
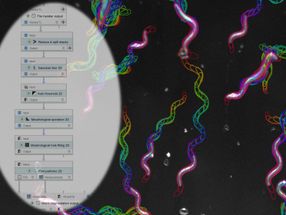
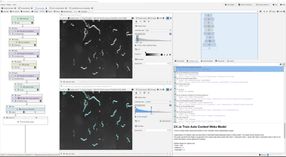
Les images, en particulier les images microscopiques, jouent un rôle majeur dans la recherche biomédicale. À l'aide de marqueurs fluorescents, par exemple, les processus dans les cellules deviennent visibles. "Une image vaut mille mots - c'est toujours vrai", déclare Thilo Figge, chef du groupe de recherche en biologie des systèmes appliquée au Leibniz-HKI et professeur à l'université Friedrich Schiller de Iéna. Mais l'analyse pose des défis croissants aux chercheurs. "Des résolutions de plus en plus élevées et donc de plus grandes quantités de données sont générées", explique Figge. "Dans le même temps, les méthodes de l'IA, ou intelligence artificielle, sont désormais si avancées qu'elles sont de plus en plus difficiles à utiliser pour les chercheurs ne possédant pas de compétences en programmation."
Le programme open source JIPipe - abréviation de Java Image Processing Pipeline - développé à Leibniz-HKI vise à simplifier cela. "JIPipe est un outil qui ne nécessite aucune compétence en programmation", explique le développeur Ruman Gerst, membre du groupe de recherche en biologie des systèmes appliqués. Le logiciel utilise plutôt un langage de programmation visuel : à l'aide de blocs de construction préfabriqués, les utilisateurs peuvent créer des flux de travail individuels pour analyser automatiquement les images en fonction de leurs besoins spécifiques.
JIPipe prend en charge d'autres langages de programmation
Le programme est basé sur le logiciel open source ImageJ, qui s'est imposé comme une norme dans l'analyse d'images scientifiques. JIPipe et ImageJ sont entièrement compatibles et se complètent dans l'analyse d'images scientifiques. "Notre programme prend en charge les scripts ImageJ et comprend les fonctions et macros habituelles", explique M. Gerst. D'autres langages de programmation tels que Python et R sont également pris en charge.
Le programme précurseur a été développé il y a plusieurs années par Zoltán Cseresnyés, également membre du groupe de recherche en biologie des systèmes appliqués. "À l'origine, j'ai écrit le code pour un test de phagocytose", explique Cseresnyés. Dans la phagocytose, une cellule prend une particule, par exemple une autre cellule, et la décompose, ce qui est généralement visualisé à l'aide de colorants fluorescents.
Au fil du temps, le spécialiste de l'imagerie a continué à étendre le code pour de nouvelles applications, et le programme est devenu lourd et trop complexe. "Nous avons réalisé que nous devions le remanier et le rendre modulaire", explique M. Cseresnyés. C'est pourquoi l'équipe a fait appel au bioinformaticien Ruman Gerst. Il a également suggéré l'utilisation du langage de programmation visuel actuel, qui permet à l'équipe d'analyser les données d'image de tout problème biomédical.
Des résultats reproductibles
JIPipe a déjà été utilisé pour plusieurs études, par exemple pour étudier l'efficacité de l'administration de médicaments par des "nanocarriers" dans le foie ou pour tester le taux de survie des nématodes qui ont digéré des bactéries productrices de toxines. Les confrontations entre les cellules immunitaires et les spores fongiques ont également été analysées avec le nouveau programme. Les développeurs proposent également des cours d'utilisation dans le cadre du Microverse Imaging Center et du NFDI4BioImage, qui fait partie de l'infrastructure nationale allemande de données de recherche. "Contrairement à l'analyse d'image manuelle, l'analyse automatisée fournit toujours les mêmes résultats, est donc reproductible et respecte les principes dits FAIR pour l'analyse d'image", souligne Thilo Figge. Le terme FAIR provient du domaine de la gestion des données de recherche et signifie"Findable, Accessible, Interoperableand Reusable".
Note: Cet article a été traduit à l'aide d'un système informatique sans intervention humaine. LUMITOS propose ces traductions automatiques pour présenter un plus large éventail d'actualités. Comme cet article a été traduit avec traduction automatique, il est possible qu'il contienne des erreurs de vocabulaire, de syntaxe ou de grammaire. L'article original dans Anglais peut être trouvé ici.