KI-Algorithmus löst Herausforderungen der Strukturbiologie
Forscher entwickeln Methoden des maschinellen Lernens, die die 3D-Formen von Arzneimittelzielen und anderen wichtigen biologischen Molekülen genau vorhersagen, selbst wenn nur wenige Daten zur Verfügung stehen
Anzeigen
Die Bestimmung der 3D-Form biologischer Moleküle ist eines der schwierigsten Probleme in der modernen Biologie und medizinischen Forschung. Unternehmen und Forschungseinrichtungen geben oft Millionen von Dollar aus, um eine Molekülstruktur zu bestimmen - und selbst solche massiven Bemühungen sind häufig erfolglos.

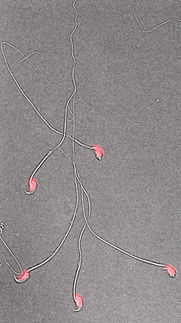
Ein neuer Algorithmus der künstlichen Intelligenz kann die 3D-Form eines RNA-Moleküls aus falschen Formen herausfiltern. Die computergestützte Vorhersage der Strukturen, in die sich RNAs falten, ist besonders wichtig - und besonders schwierig - weil so wenige Strukturen bekannt sind.
Camille L.L. Townshend
Mit Hilfe cleverer, neuer Techniken des maschinellen Lernens haben die Doktoranden Stephan Eismann und Raphael Townshend der Stanford University unter der Leitung von Ron Dror, außerordentlicher Professor für Informatik, einen Ansatz entwickelt, der dieses Problem überwindet, indem er genaue Strukturen rechnerisch vorhersagt.
Besonders bemerkenswert ist, dass ihr Ansatz selbst dann erfolgreich ist, wenn er von nur wenigen bekannten Strukturen lernt, so dass er auf die Arten von Molekülen anwendbar ist, deren Strukturen experimentell am schwierigsten zu bestimmen sind.
Ihre Arbeit wird in zwei Veröffentlichungen demonstriert, die Anwendungen für RNA-Moleküle und Multi-Protein-Komplexe beschreiben und am 27. August 2021 in Science bzw. im Dezember 2020 in Proteins veröffentlicht wurden. Der Artikel in Science ist eine Zusammenarbeit mit dem Stanford-Labor von Rhiju Das, außerordentlicher Professor für Biochemie.
"In der Strukturbiologie, die sich mit der Form von Molekülen befasst, gilt das Mantra, dass die Struktur die Funktion bestimmt", so Townshend.
Der von den Forschern entwickelte Algorithmus sagt genaue Molekülstrukturen voraus und ermöglicht es den Wissenschaftlern so, die Funktionsweise verschiedener Moleküle zu erklären.
"Proteine sind molekulare Maschinen, die alle Arten von Funktionen ausführen. Um ihre Funktionen auszuführen, binden Proteine oft an andere Proteine", so Eismann. "Wenn man weiß, dass ein Proteinpaar an einer Krankheit beteiligt ist, und wenn man weiß, wie sie in 3D interagieren, kann man versuchen, diese Interaktion mit einem Medikament ganz gezielt zu beeinflussen.
Eismann und Townshend sind gemeinsam mit dem Stanford-Postdoktoranden Andrew Watkins aus dem Das-Labor die Hauptautoren des Science-Artikels und gemeinsam mit dem ehemaligen Stanford-Doktoranden Nathaniel Thomas auch die Hauptautoren des Proteins-Artikels.
Entwurf des Algorithmus
Anstatt festzulegen, was eine Strukturvorhersage mehr oder weniger genau macht, ließen die Forscher den Algorithmus diese molekularen Merkmale selbst entdecken. Sie taten dies, weil sie feststellten, dass die herkömmliche Technik der Bereitstellung solchen Wissens einen Algorithmus zugunsten bestimmter Merkmale beeinflussen kann und ihn so daran hindert, andere informative Merkmale zu finden.
"Das Problem mit diesen handgefertigten Merkmalen in einem Algorithmus besteht darin, dass der Algorithmus sich an dem orientiert, was die Person, die diese Merkmale auswählt, für wichtig hält, und dass man möglicherweise einige Informationen verpasst, die man bräuchte, um besser abzuschneiden", so Eismann.
"Das Netzwerk hat gelernt, grundlegende Konzepte zu finden, die für die Bildung von Molekülstrukturen wichtig sind, ohne dass man es ihm ausdrücklich gesagt hat", so Townshend. "Der spannende Aspekt ist, dass der Algorithmus eindeutig Dinge gefunden hat, von denen wir wussten, dass sie wichtig sind, aber er hat auch Eigenschaften gefunden, von denen wir vorher nichts wussten."
Nach dem Erfolg mit Proteinen wandten die Forscher ihren Algorithmus als nächstes auf eine andere Klasse wichtiger biologischer Moleküle an, die RNAs. Sie testeten ihren Algorithmus in einer Reihe von "RNA-Rätseln" aus einem seit langem bestehenden Wettbewerb in ihrem Fachgebiet, und in jedem Fall übertraf das Tool alle anderen Rätselteilnehmer, und das, ohne speziell für RNA-Strukturen entwickelt worden zu sein.
Breitere Anwendungen
Die Forscher sind gespannt, wo ihr Ansatz noch angewendet werden kann, nachdem sie bereits mit Proteinkomplexen und RNA-Molekülen erfolgreich waren.
"Die meisten der dramatischen Fortschritte im maschinellen Lernen der letzten Zeit haben eine enorme Menge an Daten für das Training erfordert. Die Tatsache, dass diese Methode auch mit sehr wenigen Trainingsdaten erfolgreich ist, deutet darauf hin, dass ähnliche Methoden ungelöste Probleme in vielen Bereichen angehen könnten, in denen nur wenige Daten zur Verfügung stehen", so Dror, der Seniorautor der Proteins-Veröffentlichung und zusammen mit Das Co-Seniorautor der Science-Veröffentlichung ist.
Speziell für die Strukturbiologie ist das Team der Meinung, dass sie gerade erst an der Oberfläche des wissenschaftlichen Fortschritts kratzen, den sie erzielen können.
"Wenn man erst einmal über diese grundlegende Technologie verfügt, kann man einen weiteren Schritt im Verständnis machen und die nächsten Fragen stellen", so Townshend. "Mit diesen Informationen kann man zum Beispiel neue Moleküle und Medikamente entwerfen, ein Bereich, der die Menschen sehr begeistert."
Hinweis: Dieser Artikel wurde mit einem Computersystem ohne menschlichen Eingriff übersetzt. LUMITOS bietet diese automatischen Übersetzungen an, um eine größere Bandbreite an aktuellen Nachrichten zu präsentieren. Da dieser Artikel mit automatischer Übersetzung übersetzt wurde, ist es möglich, dass er Fehler im Vokabular, in der Syntax oder in der Grammatik enthält. Den ursprünglichen Artikel in Englisch finden Sie hier.