Composés ribosomiques d'antibiotiques décryptés
Lutte contre les bactéries multirésistantes grâce à des représentations structurelles à haute résolution
Annonces
Une équipe de recherche européenne dirigée par le département de chimie de l'université de Hambourg présente dans une étude les structures de 17 différents composés ribosomes d'antibiotiques avec une haute résolution de détails. Ces connaissances pourraient ouvrir la voie au développement de nouveaux antibiotiques pour lutter contre les bactéries multirésistantes.
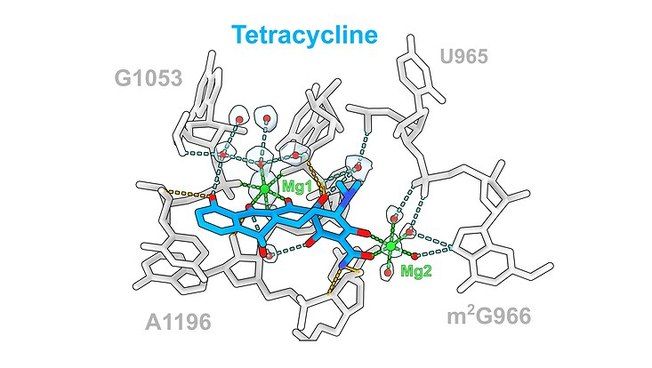
L'antibiotique tétracycline (bleu) et ses interactions avec l'ARNr ribosomique Photo : UHH/Paternoga L'antibiotique tétracycline (bleu) et ses interactions avec l'ARNr ribosomique (gris), médiées par des interactions directes (orange) ou indirectes (cyan) via Mg1 (vert) ou des molécules d'eau (rouge).
UHH/Paternoga
Les ribosomes sont de grands complexes moléculaires présents dans les cellules des plantes, des animaux, des humains ainsi que des bactéries et qui fabriquent des protéines. C'est pourquoi ils sont des cibles importantes pour les antibiotiques qui, chez les bactéries, se couplent à une sous-unité des ribosomes et provoquent des erreurs de lecture ou même l'arrêt de la lecture. Cela entraîne la formation de protéines défectueuses qui perdent leur fonction biologique, et la bactérie meurt. Cependant, de plus en plus de bactéries multirésistantes peuvent empêcher l'action des antibiotiques.
Pour contourner ces résistances, il est essentiel de bien comprendre les antibiotiques, notamment les structures des ribosomes des antibiotiques. Au cours des deux dernières décennies, des structures de ribosomes d'antibiotiques ont été publiées pour chaque classe importante d'antibiotiques dirigés vers les ribosomes, donnant un aperçu de leurs sites de liaison et de leurs mécanismes d'action.
L'équipe du professeur Daniel N. Wilson du département de chimie de l'université de Hambourg a maintenant représenté ces structures à l'aide de la cryo-microscopie électronique avec une résolution de 1,6 à 2,2 Ångström (Å), un Å correspondant à la dix millionième partie d'un millimètre. "Dans le passé, on présentait principalement des structures de ribosomes d'antibiotiques avec des résolutions de 2,5 à 3,5 Å", explique le professeur Daniel N. Wilson. "L'amélioration de la résolution nous permet maintenant d'observer même les molécules d'eau qui soutiennent les interactions entre les médicaments". Les chercheurs ont découvert qu'en général, dix à vingt liaisons hydrogène interagissaient avec le ribosome, la contribution des molécules d'eau variant selon les classes d'antibiotiques.
Les antibiotiques étudiés comprennent les six familles d'antibiotiques cliniquement pertinentes, dont les tétracyclines, utilisées pour les infections des voies respiratoires, les aminoglycosides, qui ont un large spectre d'action, entre autres pour la tuberculose ou la méningite, et les pleuromutilines, utilisées pour les infections de la peau et des tissus mous.
Pour l'étude, l'équipe de recherche a appliqué ces antibiotiques sur des ribosomes dits 70S de la bactérie "Escherichia coli" - une colibacille qui se trouve également dans l'intestin humain. "Nous pensons qu'à l'avenir, ces informations pourront être utilisées pour la conception basée sur la structure de nouveaux dérivés d'antibiotiques. En identifiant les régions qui peuvent être modifiées, l'eau liée peut être déplacée de manière ciblée et prendre ainsi le relais d'une interaction avec la cible, c'est-à-dire la bactérie", explique Wilson.
Selon Wilson, la représentation à haute résolution est un grand pas en avant : "Les anciennes structures de ribosomes d'antibiotiques avec une résolution moins élevée montrent que les sites de liaison sont similaires pour chaque classe d'antibiotiques, cependant, dans de nombreux cas, il existe des différences profondes en ce qui concerne la position exacte des substances actives ainsi que le site de liaison de la substance active". C'est pourquoi les données expérimentales à haute résolution étaient nécessaires pour obtenir une description plus précise des interactions des antibiotiques avec le ribosome.
Note: Cet article a été traduit à l'aide d'un système informatique sans intervention humaine. LUMITOS propose ces traductions automatiques pour présenter un plus large éventail d'actualités. Comme cet article a été traduit avec traduction automatique, il est possible qu'il contienne des erreurs de vocabulaire, de syntaxe ou de grammaire. L'article original dans Allemand peut être trouvé ici.










































