Cómo mapear las conexiones cerebrales usando códigos de barras de ADN
Un nuevo método desarrollado en el Laboratorio Cold Spring Harbor (CSHL) utiliza la secuenciación del ADN para trazar eficientemente las conexiones de largo alcance entre las diferentes regiones del cerebro. El enfoque reduce drásticamente el coste de mapear las conexiones de todo el cerebro en comparación con los métodos tradicionales basados en la microscopía.
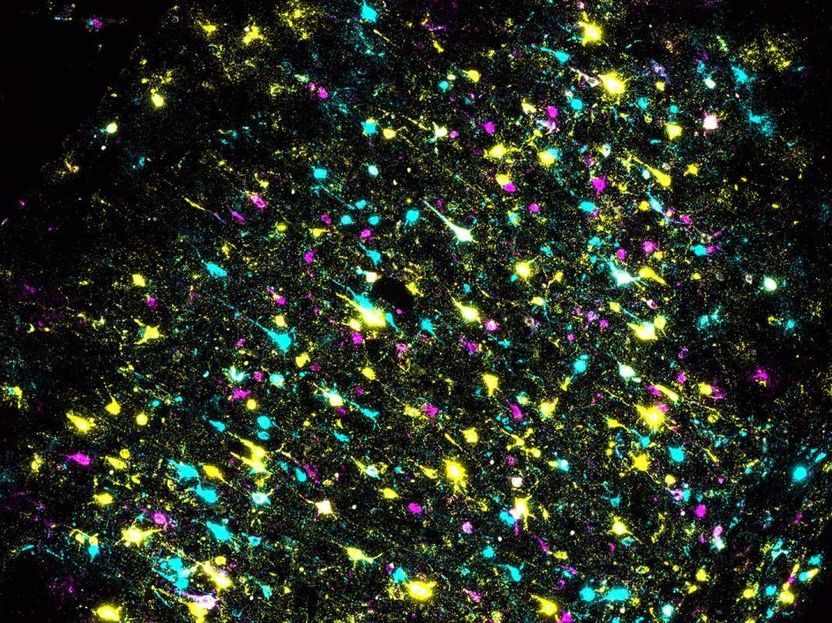
Una sección del cerebro del ratón teñida para identificar las neuronas individuales y todas sus conexiones. Cada color representa un código de barras de ADN diferente.
Xiaoyin Chen, Zador lab/CSHL
Los neurocientíficos necesitan mapas anatómicos para entender cómo fluye la información de una región del cerebro a otra. "Trazar las conexiones celulares entre las diferentes partes del cerebro -el conectoma- puede ayudar a revelar cómo el sistema nervioso procesa la información, así como cómo los cables defectuosos contribuyen a las enfermedades mentales y otros trastornos", dice Longwen Huang, investigador postdoctoral en el laboratorio del profesor Anthony Zador del CSHL. La creación de estos mapas ha sido costosa y ha llevado mucho tiempo, exigiendo esfuerzos masivos que están fuera del alcance de la mayoría de los equipos de investigación.
Los investigadores suelen seguir las trayectorias de las neuronas utilizando etiquetas fluorescentes, que pueden destacar cómo las células individuales se ramifican a través de una red neuronal enmarañada para encontrar y conectar con sus objetivos. Pero, la paleta de etiquetas fluorescentes adecuadas para este trabajo es limitada. Los investigadores pueden inyectar tintes de diferentes colores en dos o tres partes del cerebro, y luego rastrear las conexiones que emanan de esas regiones. Pueden repetir este proceso, apuntando a nuevas regiones, para visualizar conexiones adicionales. Para generar un mapa de todo el cerebro, esto debe hacerse cientos de veces, usando nuevos animales de investigación cada vez.
El método desarrollado en el laboratorio de Zador, llamado Secuenciación de Conectomas Individuales de Animales en Todo el Cerebro (BRICseq), tiene un enfoque diferente. "Ya no etiquetamos las regiones del cerebro y sus proyecciones usando colores. Las etiquetamos usando secuencias de nucleótidos", dice Huang. Combinando las cuatro letras del código de ADN en breves "códigos de barras" se genera un número virtualmente infinito de etiquetas que pueden distinguir una célula de otra, explica. Después de etiquetar, los investigadores utilizan la secuenciación del ADN para analizar segmentos diminutos de tejido cerebral, interpretando cada código de barras recurrente como una señal de una conexión celular.
"La diversidad de los códigos de barras es realmente alta comparada con el número de colores que podemos usar en la investigación científica. Así que ahora podemos etiquetar realmente una enorme cantidad de neuronas y regiones cerebrales por animal, lo que nos permite mapear las proyecciones de múltiples regiones cerebrales utilizando estos códigos de barras", dice Huang.
El equipo de investigación, incluyendo al ex-estudiante de postgrado Justus Kebschull, que trabajó con Huang para desarrollar la técnica, informa en la revista Cell que el BRICseq mapea con precisión la conectividad de región a región en los cerebros de los ratones. Dicen que el enfoque será ampliamente accesible y debería ser adaptable a otros organismos.
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.