ARN en acción: filmación del autoensamblaje de una ribozima
Los investigadores han visualizado, con un detalle sin precedentes, cómo una gran molécula de ARN se ensambla en una máquina funcional
El ARN es una molécula biológica fundamental, hoy ampliamente aprovechada en medicina y nanotecnología. Al igual que las proteínas, el ARN suele obtener su función de su estructura tridimensional. Un reciente estudio publicado en Nature Communications ha captado por primera vez una ribozima en movimiento, casi fotograma a fotograma. Los investigadores grabaron cómo esta diminuta máquina de ARN se pliega, flexiona y ensambla, revelando su intrincada coreografía con un detalle sin precedentes.
Utilizando las técnicas más avanzadas -criomicroscopía electrónica (criomicroscopía electrónica), dispersión de rayos X de ángulo pequeño (SAXS), bioquímica y enzimología del ARN, procesamiento de imágenes y simulaciones moleculares-, los científicos observaron el ensamblaje de una ribozima autoempalmante, una molécula de ARN que puede "cortar y pegar" su propia secuencia, editándose a sí misma para volverse operativa. Captaron el proceso dinámico "entre bastidores" por el que la ribozima autoempalmante se pliega en su estructura funcional. La investigación fue dirigida por el equipo de Marco Marcia , antiguo jefe de grupo del EMBL y actual profesor asociado y jefe de grupo de SciLifeLab en la Universidad de Uppsala (Suecia).
Este avance fue posible gracias a las instalaciones de vanguardia y a los servicios especializados del EMBL de Grenoble, que permitieron integrar métodos avanzados de biología estructural con la bioquímica y la enzimología del ARN. El grupo de Marcia también se benefició de la estrecha colaboración con el Centro de Biología de Sistemas Estructurales (CSSB) de Hamburgo, donde se desarrollaron enfoques innovadores de procesamiento de imágenes crio-EM adaptados a este proyecto específico, y con el Istituto Italiano di Tecnologia (IIT), que aportó conocimientos de simulación molecular de alto nivel.
"Determinar las estructuras del ARN es una tarea difícil: la flexibilidad inherente y la carga negativa hacen del ARN un objetivo notoriamente difícil para los estudios estructurales", dijo Shekhar Jadhav, antiguo becario predoctoral en el EMBL de Grenoble, ahora postdoctorado en la Universidad de Uppsala, Suecia. "Los esfuerzos persistentes y el cribado exhaustivo en microscopios electrónicos nos llevaron finalmente a visualizar la esquiva dinámica del ARN".

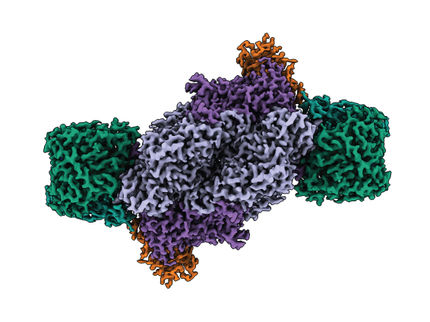
La imagen representa mapas de densidad crio-EM para los dos estados conformacionales extremos que el intrón del grupo II adquiere durante el plegamiento, a través de un movimiento dinámico continuo de sus motivos helicoidales estructurados.
Shekhar Jadhav/EMBL
Cómo un dominio orquesta la trama del ARN
En el corazón de esta producción se encuentra el Dominio 1 (D1), el andamiaje central de la ribozima y, según parece, su director. Este dominio actúa como una puerta molecular, indicando a los otros dominios (D2, D3, D4) que entren en el momento preciso durante el proceso de plegamiento.
Movimientos sutiles en partes clave de la molécula D1 hacen que una de sus secciones se abra y deje paso a la siguiente. Cada dominio se une a la escena sólo cuando el anterior está correctamente colocado, creando una secuencia perfecta de coreografía molecular que evita errores estructurales y garantiza un final impecable: la formación de una estructura que puede catalizar una reacción química, esencial para la función de la ribozima.
Captar las tomas ocultas
Analizando cientos de miles de partículas individuales de ARN, el equipo reconstruyó "tomas" intermedias que eran invisibles en las estructuras cristalinas estáticas. Estos fotogramas fugaces muestran cómo el ARN explora posturas alternativas antes de establecerse en su conformación final.
Representación dinámica de mapas de densidad para los dos estados conformacionales extremos que adquiere el intrón del grupo II durante el plegamiento, a través de un movimiento dinámico continuo de sus motivos helicoidales estructurados. Shekhar Jadhav/EMBL
"Para captar estos fotogramas fugaces, tuvimos que desarrollar novedosas estrategias de procesamiento de imágenes crio-EM", afirma Maya Topf , jefa de grupo en CSSB , profesora del Centro Médico Universitario de Hamburgo-Eppendorf y colaboradora en el estudio. "Este es un gran ejemplo de cómo la innovación computacional y los datos crioEM de alta calidad pueden revelar las conformaciones ocultas de las máquinas moleculares".
Los datos SAXS y las simulaciones de dinámica molecular ayudaron a los científicos a perfeccionar cada fotograma y montar el argumento.
"Uno de los principales puntos fuertes de este trabajo es la sinergia entre estos nuevos datos estructurales de vanguardia sobre el ARN y nuestras avanzadas simulaciones moleculares de este difícil sistema", afirma Marco De Vivo , Jefe del Laboratorio de Modelado Molecular y Descubrimiento de Fármacos y Director Asociado de Computación del Institu Italiano di Technologia de Génova, y uno de los colaboradores en este estudio. "Este enfoque combinado ha aclarado, con un nivel de detalle atomístico sin precedentes, la dinámica que impulsa todo el ensamblaje de esta molécula de ARN, lo que ahora abre nuevas vías para los esfuerzos de descubrimiento de fármacos dirigidos al ARN."
De los guiones antiguos a los derivados modernos
Se cree que los intrones del grupo II, las ribozimas que aparecen en esta película molecular, son los antepasados del espliceosoma, la compleja maquinaria que edita el ARN en las células humanas. Al revelar cómo estas moléculas se pliegan eficazmente y evitan las trampas cinéticas, el estudio aporta nuevos conocimientos sobre cómo la vida primitiva basada en el ARN pudo haber desarrollado sus herramientas de edición. Más allá de la tradición evolutiva, este trabajo también prepara el terreno para el diseño y la ingeniería del ARN, orientando el modo en que las futuras biotecnologías podrían programar las moléculas de ARN para que se plieguen correctamente con fines terapéuticos o nanobiotecnológicos.
Abrir la puerta a la inteligencia artificial del ARN
Los detallados conjuntos de datos y mecanismos moleculares descubiertos en este estudio ofrecen un valioso punto de referencia para entrenar y probar modelos de IA. Algunas de las estructuras de ARN resueltas aquí ya se han utilizado en concursos internacionales CASP -el mismo reto de predicción que dio origen a AlphaFold-, como se ha descrito recientemente en la revista Proteins .
"Se espera que este trabajo desempeñe un papel clave en la configuración de enfoques de inteligencia artificial para la predicción de estructuras de ARN, allanando el camino hacia un nuevo 'AlphaFold para el ARN'", afirmó Marcia.
Esta convergencia de precisión experimental y aprendizaje automático marca una nueva fase para la biología estructural del ARN, en la que la IA y la crioelectrónica pueden aprender la una de la otra para predecir, visualizar y comprender la dinámica de la molécula más versátil de la vida".
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.