Etichettatura delle proteine più rapida e luminosa con il nuovo strumento SNAP-tag2
Il nuovo sistema di etichettatura funziona efficacemente in un'ampia gamma di applicazioni di bioimaging e apre nuove possibilità per l'imaging multicolore
Annunci
La proteina SNAP-tag è un potente strumento per etichettare le proteine con fluorofori sintetici per il bioimaging. Gli scienziati del Max Planck Institute for Medical Research di Heidelberg hanno messo a punto una versione molto migliorata, denominata SNAP-tag2, nonché substrati ottimizzati per un'etichettatura più rapida nelle cellule vive.
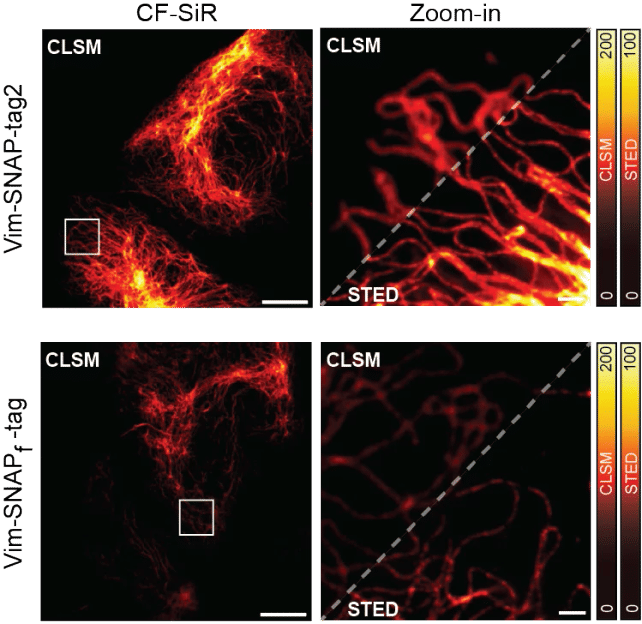
Rispetto allo SNAP-tag comunemente utilizzato, lo SNAP-tag2 offre una capacità di etichettatura notevolmente migliorata utilizzando tecniche di imaging in microscopia a super-risoluzione come la microscopia STED (immagini a destra).
© MPI for Medical Research
Un mutante SNAP-tag altamente ingegnerizzato
I tag proteici auto-etichettati, come SNAP-tag, offrono l'opportunità di attaccare fluorofori sintetici luminosi e fotostabili alle proteine di interesse per una successiva analisi e sono ampiamente utilizzati in biochimica. ampiamente utilizzati in biochimica.
"SNAP-tag è stato originariamente sviluppato a partire da un enzima umano di riparazione del DNA. L'ulteriore ingegnerizzazione di una proteina già pesantemente modificata è stata una delle sfide più grandi", afferma Stefanie Kühn, che ha guidato il team del progetto composto da altri scienziati del Max Planck Institute (MPI) per la ricerca medica, tra cui Julien Hiblot, Veselin Nasufovic e il direttore Kai Johnsson, e da un team dell'Università di Groningen. "Inoltre, volevamo trovare un nucleo di substrato che funzionasse bene nelle cellule con vari fluorofori attaccati. Abbiamo deciso di utilizzare una combinazione di ottimizzazione del substrato e ingegneria proteica per sviluppare SNAP-tag2".
Maggiore reattività
L'utilità dello SNAP-tag, finora ampiamente utilizzato nelle applicazioni a cellule vive, può essere limitata dalla sua cinetica di etichettatura relativamente lenta e dalla limitata permeabilità cellulare dei suoi substrati. Gli scienziati dell'MPI per la ricerca medica volevano trovare una soluzione a questo problema. SNAP-tag2 reagisce rapidamente con i nuovi substrati permeabili alle cellule: Mostra una costante di velocità di etichettatura che corrisponde a un miglioramento di 100 volte rispetto a coppie SNAP-tag-substrato comparabili.
Etichettatura di fluorescenza più efficiente in cellule vive
SNAP-tag2 offre ulteriori vantaggi. Quando viene etichettato con coloranti altamente fluorogenici, mostra anche un aumento di 5 volte della luminosità della fluorescenza rispetto allo SNAP-tag attualmente utilizzato. La cinetica di etichettatura e la luminosità più rapide di SNAP-tag2 si traducono in prestazioni notevolmente migliorate nelle cellule di mammifero e nel lievito. Consente inoltre di utilizzare tecniche di imaging di microscopia a super-risoluzione come la microscopia STED e di lavorare con altri tipi di cellule notoriamente refrattarie all'etichettatura chimica, come le cellule di lievito.
"SNAP-tag2, insieme ai suoi substrati migliorati, è superiore alle versioni di SNAP-tag precedentemente utilizzate in tutte le applicazioni che abbiamo testato e speriamo che possa eccellere anche nelle applicazioni in vivo ", afferma Stefanie Kühn.
Possibili altre aree di applicazione
In sintesi, il team prevede che i miglioramenti apportati a SNAP-tag2 faranno progredire l'utilità di questo strumento, già ampiamente adottato, per l'imaging di cellule vive e per altre applicazioni nelle scienze biologiche. Inoltre, SNAP-tag2 apre nuove possibilità per le applicazioni di imaging multicolore.
Nota: questo articolo è stato tradotto utilizzando un sistema informatico senza intervento umano. LUMITOS offre queste traduzioni automatiche per presentare una gamma più ampia di notizie attuali. Poiché questo articolo è stato tradotto con traduzione automatica, è possibile che contenga errori di vocabolario, sintassi o grammatica. L'articolo originale in Inglese può essere trovato qui.
Pubblicazione originale
Stefanie Kühn, Veselin Nasufovic, Jonas Wilhelm, Julian Kompa, Eline M. F. de Lange, Yin-Hsi Lin, Cornelia Egoldt, Jonas Fischer, Artem Lennoi, Miroslaw Tarnawski, Jochen Reinstein, Rifka Vlijm, Julien Hiblot, Kai Johnsson; "SNAP-tag2 for faster and brighter protein labeling"; Nature Chemical Biology, 2025-7-3























































