Une nouvelle méthode révolutionne le diagnostic du cancer
La technologie révolutionnaire "Deep Visual Proteomics" réunit quatre technologies
Comment le cancer apparaît-il ? Comment la composition cellulaire influence-t-elle la malignité des tumeurs ? Ces questions sont profondes et difficiles à résoudre, mais elles sont cruciales pour comprendre la maladie et trouver le bon traitement. Aujourd'hui, une équipe germano-danoise dirigée par le professeur Matthias Mann a mis au point une technologie révolutionnaire appelée "Deep Visual Proteomics". Cette méthode fournit aux chercheurs et aux cliniciens une lecture des protéines pour comprendre le cancer à une résolution de type cellulaire unique. La technologie a été publiée dans la revue Nature Biotechnology et démontre son potentiel dans une première application aux cellules cancéreuses.
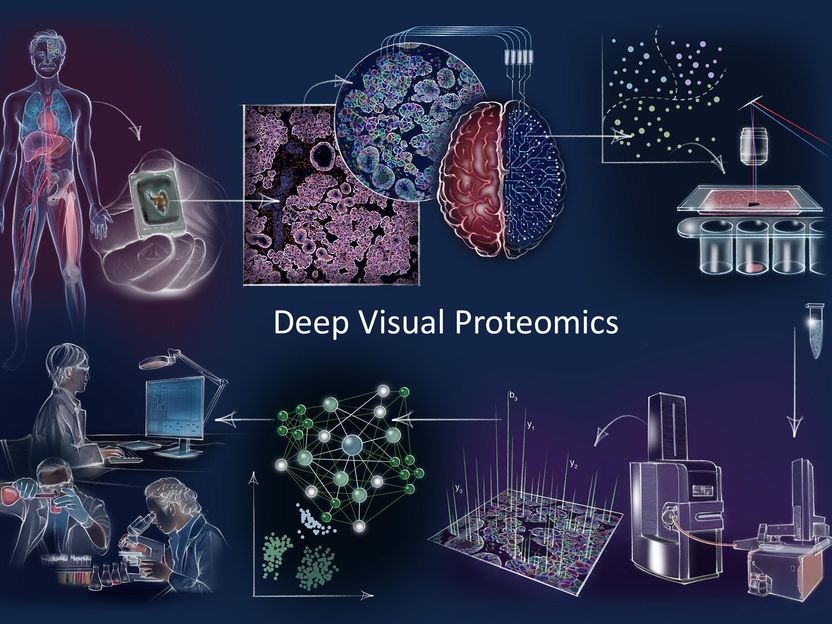
Concept et flux de travail de Deep Visual Proteomics Dans le sens des aiguilles d'une montre : La Deep Visual Proteomics (DVP) combine l'imagerie à haute résolution, l'analyse d'images guidée par l'intelligence artificielle (IA) pour la classification et l'isolement de cellules uniques avec un nouveau flux de travail protéomique ultra-sensible. La DVP associe une imagerie riche en données de cultures cellulaires ou de tissus archivés de biobanques de patients à une segmentation cellulaire basée sur l'apprentissage profond et à une identification des types et des états cellulaires basée sur l'apprentissage automatique. Les objets cellulaires ou subcellulaires d'intérêt classés par l'IA (non) supervisée subissent une microdissection laser automatisée et un profilage protéomique basé sur la spectrométrie de masse (MS). L'analyse ultérieure des données bioinformatiques permet l'exploration des données pour découvrir des signatures protéiques fournissant des informations moléculaires sur la variation du protéome dans les états de santé et de maladie au niveau des cellules individuelles.
© MPI of Biochemistry
Les protéines sont parmi les acteurs les plus importants dans une variété de maladies. Qualifiées à juste titre de "bêtes de somme moléculaires de la cellule", leur bon fonctionnement détermine souvent la santé d'une cellule et, par extension, celle d'un individu.
Matthias Mann explique : "Lorsque quelque chose ne va pas à l'intérieur de nos cellules et que nous tombons malades, vous pouvez être sûr que les protéines sont impliquées de nombreuses manières différentes. Pour cette raison, la cartographie du paysage protéique peut nous aider à déterminer pourquoi une tumeur peut se développer chez un patient particulier, quelles sont les vulnérabilités de cette tumeur et aussi quelle stratégie de traitement pourrait s'avérer la plus bénéfique."
Intriguée par ces questions, une équipe de scientifiques pluridisciplinaires des groupes de recherche dirigés par Matthias Mann à l'Institut Max Planck (MPI) de biochimie, près de Munich, et au Centre de recherche sur les protéines (CPR) de la Fondation Novo Nordisk à l'université de Copenhague, a inventé une méthode innovante appelée "Deep Visual Proteomics". L'étude combine les caractéristiques visuelles d'une tumeur avec une technologie de profilage profond pour visualiser les signatures protéiques dans les cellules aberrantes qui sont adjacentes aux cellules saines environnantes. Les chercheurs obtiendront ainsi un aperçu sans précédent de la maladie et guideront les oncologues dans l'élaboration de stratégies intelligentes de diagnostic et de traitement.
Deep Visual Proteomics réunit quatre technologies
Deep Visual Proteomics intègre pour la première fois les avancées de quatre technologies différentes en un seul flux de travail : Premièrement, la microscopie avancée génère des cartes tissulaires à haute résolution. Ensuite, des algorithmes d'apprentissage automatique et d'intelligence artificielle (IA) sont utilisés pour classer avec précision les cellules en fonction de leur forme, de leur taille ou de la localisation des protéines avant que les cellules individuelles ne soient collectées par microdissection par capture laser de haute précision. Ensuite, après avoir trié les cellules normales ou différents types de cellules malades, les milliers de protéines présentes dans les populations cellulaires sont détectées en une seule fois à l'aide d'instruments de spectrométrie de masse (MS) ultra-sensibles à partir de cette quantité infime d'échantillon. Enfin, des analyses bioinformatiques sophistiquées génèrent des cartes de protéines qui fournissent une résolution spatiale des signatures protéiques dans des maladies très complexes comme le cancer. Ces paysages protéiques sont des outils précieux pour les cliniciens, qui peuvent ainsi mieux comprendre les mécanismes de la santé et de la maladie.
Notre nouveau concept, la "protéomique visuelle profonde", pourrait changer la donne en matière de pathologie moléculaire dans les hôpitaux. Avec cette méthode, nous prenons un échantillon de tissu avec des cellules tumorales, et nous pouvons identifier et déterminer des milliers de protéines en une minute de temps et d'effort. Ces signatures protéomiques révèlent les mécanismes de développement des tumeurs et exposent directement de nouvelles cibles thérapeutiques à partir d'une seule tranche de tissu d'une biopsie de patient cancéreux. Elle expose un cosmos de molécules à l'intérieur de ces cellules cancéreuses", déclare Andreas Mund, professeur associé au CPR et membre de l'équipe du professeur Matthias Mann, qui a été à l'origine de ce développement.
Pertinence pour la pathologie clinique
Dans cette nouvelle étude, les chercheurs ont appliqué la "protéomique visuelle profonde" à des cellules provenant de patients atteints de carcinome des cellules acineuses et de mélanome. Lise Mette Rahbek Gjerdrum, consultante et professeur associé de recherche clinique au département de pathologie de l'hôpital universitaire de Zélande et au département de médecine clinique de l'université de Copenhague, décrit : "Cette méthode unique combine l'architecture des tissus avec l'expression de milliers de protéines spécifiques à des cellules sélectionnées. Elle permet aux chercheurs d'étudier les interactions entre les cellules cancéreuses et les cellules environnantes, avec des implications majeures pour le futur traitement clinique du cancer. Récemment, nous avons diagnostiqué un cas clinique très complexe à l'aide de la "Deep Visual Proteomics".
Le Dr Fabian Coscia, coauteur principal de l'étude et - depuis juin 2021 - chef du groupe de recherche "Protéomique spatiale" au Centre Max Delbrück de médecine moléculaire de l'Association Helmholtz (MDC) à Berlin, déclare : "Mais bien sûr, la technique peut être utilisée pour caractériser tous les autres types de tumeurs avec des détails similaires." Son objectif, dit-il, est d'exploiter les biobanques de patients archivées et de trouver de nouvelles cibles pour des thérapies anticancéreuses individualisées, c'est-à-dire adaptées au patient - même pour des tumeurs auparavant résistantes aux traitements.
La "protéomique visuelle profonde" ne permet pas seulement de mieux comprendre le cancer. La méthode peut également être appliquée à d'autres maladies. "Par exemple, vous pouvez analyser les protéines des cellules nerveuses pour découvrir exactement ce qui se passe dans les cellules dans les maladies neurodégénératives telles que la maladie d'Alzheimer ou de Parkinson", explique encore Fabian Coscia.
''En fusionnant la puissance de la microscopie, de l'IA et de la protéomique ultra-sensible basée sur la MS, nous avons développé une méthode très puissante pour comprendre le câblage moléculaire des cellules saines par rapport aux cellules malades. Cela pourrait aider les médecins à identifier des cibles pour les futurs médicaments et diagnostics'', conclut Matthias Mann.
Note: Cet article a été traduit à l'aide d'un système informatique sans intervention humaine. LUMITOS propose ces traductions automatiques pour présenter un plus large éventail d'actualités. Comme cet article a été traduit avec traduction automatique, il est possible qu'il contienne des erreurs de vocabulaire, de syntaxe ou de grammaire. L'article original dans Anglais peut être trouvé ici.
Publication originale
A. Mund*#, F. Coscia*, A. Kriston, R. Hollandi, F. Kovács, A.-D. Brunner, E. Migh, L. Schweizer, A. Santos Delgado, M. Bzorek, S. Naimy, L.M. Rahbek Gjerdrum7,13, B. Dyring-Andersen, J. Bulkescher, C. Lukas, M.A. Eckert, E. Lengyel, C. Gnann, E. Lundberg, P. Horvath#, M. Mann#; * shared first-authors and # corresponding authors; Deep Visual Proteomics defines cell identity and heterogeneity; Nature Biotechnology, May 2022





































