L'abondance de "portes secrètes" sur les protéines humaines pourrait révolutionner la découverte de médicaments
L'identification de vulnérabilités cachées à la surface de protéines "indéracinables" pourrait transformer le traitement des maladies
Le nombre de cibles thérapeutiques potentielles à la surface des protéines humaines est beaucoup plus important qu'on ne le pensait, selon les conclusions d'une nouvelle étude publiée dans la revue Nature.
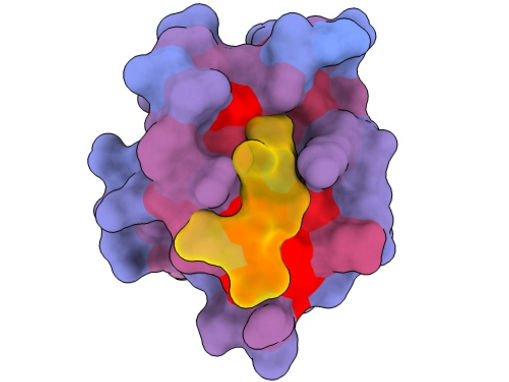
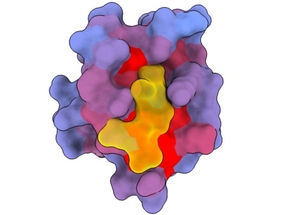
Une image tridimensionnelle montrant la protéine humaine PSD95-PDZ3 sous différents angles. On voit le partenaire de liaison CRIPT (jaune) dans le site actif, le gradient de couleur bleu-rouge indiquant un potentiel croissant d'effets allostériques. Basé sur l'accession PDB 1BE9.
André Faure/CRG
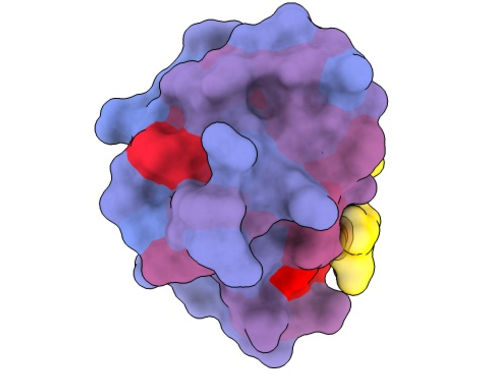
Une image tridimensionnelle montrant la protéine humaine PSD95-PDZ3 sous différents angles. On voit le partenaire de liaison CRIPT (jaune) dans le site actif, le gradient de couleur bleu-rouge indiquant un potentiel croissant d'effets allostériques. Basé sur l'accession PDB 1BE9.
André Faure/CRG
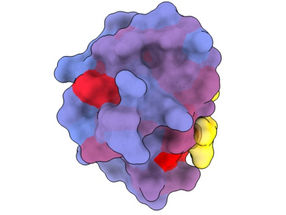
Une nouvelle technique révolutionnaire mise au point par des chercheurs du Centre de régulation génomique (CRG) de Barcelone a révélé l'existence d'une multitude de portes jusqu'alors secrètes qui contrôlent la fonction des protéines et qui pourraient, en théorie, être ciblées pour changer radicalement le cours de pathologies aussi variées que la démence, le cancer et les maladies infectieuses.
La méthode, dans laquelle des dizaines de milliers d'expériences sont réalisées en même temps, a été utilisée pour dresser la toute première carte de ces cibles insaisissables, également appelées sites allostériques, dans deux des protéines humaines les plus courantes, révélant qu'elles sont abondantes et identifiables.
Cette approche pourrait changer la donne en matière de découverte de médicaments, et permettre de mettre au point des médicaments plus sûrs, plus intelligents et plus efficaces. Elle permet aux laboratoires de recherche du monde entier de trouver et d'exploiter les vulnérabilités de n'importe quelle protéine, y compris celles que l'on pensait jusqu'à présent "inattaquables".
"Non seulement ces sites thérapeutiques potentiels sont abondants, mais il est prouvé qu'ils peuvent être manipulés de nombreuses manières différentes. Plutôt que de simplement les allumer ou les éteindre, nous pourrions moduler leur activité comme un thermostat. Du point de vue de l'ingénierie, c'est de l'or en barre, car cela nous donne toute latitude pour concevoir des "médicaments intelligents" qui ciblent le mauvais et épargnent le bon", explique André Faure, chercheur postdoctoral au CRG et coauteur principal de l'article.
Les protéines jouent un rôle central dans tous les organismes vivants et remplissent des fonctions vitales telles que fournir une structure, accélérer les réactions, servir de messagers ou combattre les maladies. Elles sont constituées d'acides aminés qui se plient en d'innombrables formes différentes dans l'espace tridimensionnel. La forme d'une protéine est cruciale pour sa fonction, une seule erreur dans une séquence d'acides aminés pouvant avoir des conséquences dévastatrices pour la santé humaine.
L'allostérie est l'un des grands mystères non résolus de la fonction des protéines. Les effets allostériques se produisent lorsqu'une molécule se lie à la surface d'une protéine, ce qui provoque à son tour des changements sur un site distant de la même protéine, régulant ainsi sa fonction à distance. De nombreuses mutations à l'origine de maladies, dont de nombreux facteurs de cancer, sont pathologiques en raison de leurs effets allostériques.
Malgré leur importance fondamentale, les sites allostériques sont incroyablement difficiles à trouver. En effet, les règles qui régissent le fonctionnement des protéines au niveau atomique sont cachées à l'abri des regards. Par exemple, une protéine peut se transformer en présence d'une molécule entrante, révélant des poches cachées au plus profond de sa surface, potentiellement allostériques mais non identifiables par la seule détermination de la structure conventionnelle.
Les chasseurs de médicaments ont traditionnellement conçu des traitements qui ciblent le site actif d'une protéine, la petite région où les réactions chimiques se produisent ou les cibles sont liées. L'inconvénient de ces médicaments, également appelés médicaments orthostériques, est que les sites actifs de nombreuses protéines se ressemblent beaucoup et que les médicaments ont donc tendance à se lier et à inhiber de nombreuses protéines différentes en même temps, ce qui entraîne des effets secondaires potentiels. En comparaison, la spécificité des sites allostériques signifie que les médicaments allostériques font partie des types de médicaments les plus efficaces actuellement disponibles. De nombreux médicaments allostériques, qui traitent diverses pathologies allant du cancer au SIDA en passant par les troubles hormonaux, ont été découverts par hasard.
Les auteurs de l'étude ont relevé ce défi en développant une technique appelée double ACP profonde (ddPCA), qu'ils décrivent comme une "expérience de force brute". "Nous cassons délibérément les choses de milliers de façons différentes pour construire une image complète de la façon dont quelque chose fonctionne", explique le professeur de recherche de l'ICREA Ben Lehner, coordinateur du programme de biologie des systèmes au CRG et auteur de l'étude. "C'est comme si l'on soupçonnait une bougie d'allumage défectueuse, mais qu'au lieu de se contenter de la vérifier, le mécanicien démonte toute la voiture et la contrôle pièce par pièce. En testant dix mille choses en une seule fois, nous identifions toutes les pièces qui comptent vraiment."
La méthode fonctionne en modifiant les acides aminés qui composent une protéine, ce qui donne des milliers de versions différentes de la protéine avec seulement une ou deux différences dans la séquence. Les effets des mutations sont ensuite testés en même temps dans des cellules vivantes en laboratoire.
"Chaque cellule est une minuscule usine qui fabrique une version différente de la protéine. Dans un seul tube à essai, nous avons des millions d'usines différentes et nous pouvons donc tester très rapidement le fonctionnement de toutes les différentes versions d'une protéine", ajoute le Dr Lehner. Les données recueillies au cours des expériences sont introduites dans des réseaux neuronaux, des algorithmes qui analysent les données en imitant le fonctionnement du cerveau humain, ce qui permet d'établir des cartes complètes indiquant l'emplacement des sites allostériques à la surface des protéines.
L'un des grands avantages de cette méthode est qu'il s'agit d'une technique abordable, accessible à tout laboratoire de recherche dans le monde. "Elle simplifie considérablement le processus nécessaire à la recherche de sites allostériques et offre un niveau de précision supérieur à celui de plusieurs autres méthodes de laboratoire plus coûteuses et plus longues", explique Júlia Domingo, coauteur de l'étude. "Notre espoir est que d'autres scientifiques utilisent cette technique pour cartographier rapidement et de manière exhaustive les sites allostériques des protéines humaines, un par un."
L'un des avantages à plus long terme de la technique est son potentiel pour étudier la fonction et l'évolution des protéines. Les auteurs de l'étude estiment que, si elle est mise à l'échelle, la méthode pourrait un jour déboucher sur des avancées permettant de prédire précisément les propriétés des protéines à partir de leurs séquences d'acides aminés. En cas de succès, les auteurs affirment que cela ouvrirait une nouvelle ère de biologie moléculaire prédictive, permettant un développement beaucoup plus rapide de nouveaux médicaments et d'une industrie propre, basée sur la biologie.
"Alors que certains outils peuvent prédire la structure d'une protéine en lisant sa séquence, notre méthode va plus loin en nous indiquant comment une protéine fonctionne. Elle s'inscrit dans une vision plus large visant à rendre la biologie aussi facile à concevoir que les avions, les ponts ou les ordinateurs. Nous sommes confrontés aux mêmes défis depuis plus de 70 ans, mais il s'avère qu'ils sont plus faciles à relever que nous le pensions. Si nous réussissons, cela ouvrira un nouveau domaine aux possibilités sans précédent", conclut le Dr Lehner.
Note: Cet article a été traduit à l'aide d'un système informatique sans intervention humaine. LUMITOS propose ces traductions automatiques pour présenter un plus large éventail d'actualités. Comme cet article a été traduit avec traduction automatique, il est possible qu'il contienne des erreurs de vocabulaire, de syntaxe ou de grammaire. L'article original dans Anglais peut être trouvé ici.