Des chercheurs de l'Université de Graz ouvrent de nouvelles voies pour comprendre les protéines
Une méthode récemment mise au point, qui compare les séquences de protéines générées par l'IA aux séquences naturelles, permet de déterminer avec beaucoup plus de précision qu'auparavant les acides aminés qui régulent la fonction et la structure.
Les protéines font partie des éléments constitutifs les plus importants de la nature et jouent un rôle central dans les processus biologiques de tous les organismes. C'est pourquoi les scientifiques souhaitent les comprendre le plus précisément possible. En tant que polymères de différents acides aminés, les protéines peuvent avoir différentes structures tridimensionnelles et diverses fonctions. Cependant, il est souvent difficile de déterminer quels acides aminés influencent la fonction de la protéine et lesquels influencent la stabilité structurelle.
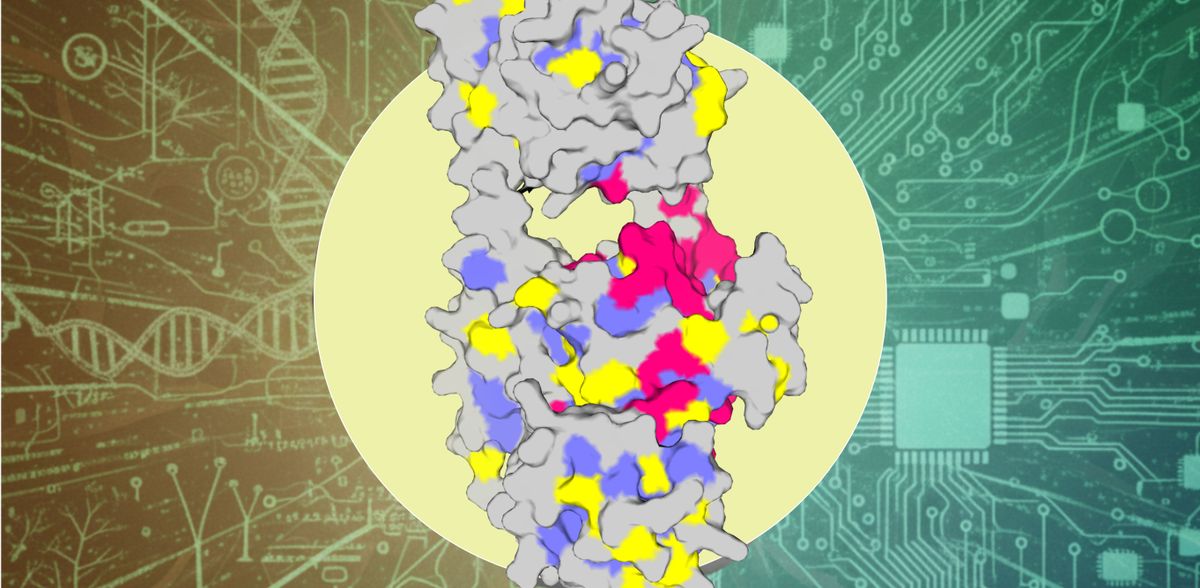
Une équipe dirigée par Andreas Winkler et Oliver Eder de l'Institut de biochimie de l'Université de technologie de Graz (TU Graz) a réalisé une percée en utilisant l'approche dite Fonction-Structure-Adaptabilité (FSA), qu'elle vient de publier dans la revue "Structure". FSA compare des séquences de protéines idéales générées par apprentissage automatique avec des séquences naturelles qui se sont développées au cours de millions d'années d'évolution. Cela permet d'identifier avec une précision sans précédent les acides aminés qui sont essentiels à la fonction et à la stabilité. Ces connaissances constituent une base importante pour la production et la modification des protéines et donc pour le développement de nouveaux médicaments, pour l'amélioration ciblée des protéines dans les applications industrielles et pour une meilleure compréhension des modifications des protéines, par exemple en relation avec la résistance aux antibiotiques.
Mieux comprendre les éléments constitutifs de la vie
"En tant que biochimistes, nous voulons comprendre comment les protéines ont évolué dans la nature et découvrir quels acides aminés sont importants pour des fonctions spécifiques", explique Andreas Winkler. "Pour ce faire, nous avons combiné ce que la nature a conservé au cours de l'évolution avec ce qu'un modèle d'intelligence artificielle considère comme pertinent pour la stabilité et la structure d'une protéine. Cette combinaison de millions d'années d'histoire de l'évolution et de technologies de pointe simplifie grandement l'analyse et la compréhension des protéines".
Pour sa méthode, l'équipe a utilisé le modèle d'apprentissage profond ProteinMPNN, qui génère de nouvelles séquences de protéines en veillant à ce qu'elles adoptent une structure tridimensionnelle stable prédéterminée. Les chercheurs ont comparé ces séquences avec celles des protéines naturelles. La famille de protéines bactériophytochromes a été utilisée comme système de test. Dans la nature, elle sert de photorécepteur à certaines bactéries et joue un rôle central dans la perception des influences de l'environnement telles que la lumière. La nouvelle méthode d'analyse a révélé que si un acide aminé est représenté à plusieurs reprises dans les séquences naturelles, mais ne semble pas significatif pour la protéineMPNN, cela indique un rôle fonctionnel. En revanche, s'il est fortement présent dans les deux collections de séquences, cela indique une importance structurelle.
Validation en laboratoire
Pour leur approche, les chercheurs ont dû regrouper les acides aminés sur la base de propriétés chimiques afin de pouvoir ensuite comparer statistiquement les protéines naturelles et celles générées par l'IA. Cela a permis de classer les acides aminés en trois catégories : "fonctionnel" (important pour le rôle spécifique de la protéine), "structurel" (important pour la stabilité et le repliement) et "adaptable" (une troisième catégorie qui nécessite encore des recherches plus approfondies). L'équipe a validé les résultats au moyen d'expériences de laboratoire approfondies au cours desquelles elle a pu influencer les propriétés fonctionnelles des protéines en apportant des modifications spécifiques aux acides aminés classés dans les catégories correspondantes. Il a ainsi été possible, par exemple, d'influencer de manière significative la perception de la lumière par le système de test des photorécepteurs. La comparaison avec des résidus fonctionnels déjà connus dans la littérature a également confirmé le taux de réussite élevé de la nouvelle méthode d'analyse.
"Dans le passé, il fallait souvent plusieurs mois, voire plusieurs années de travail préparatoire et de laboratoire pour réaliser une analyse de ce type", explique Oliver Eder. "Le travail préliminaire d'identification des séquences de protéines naturelles potentiellement intéressantes est désormais possible pour une nouvelle protéine en une semaine. Et comme notre méthode nous permet de pré-filtrer les acides aminés fonctionnels de manière beaucoup plus spécifique, nous n'avons pas besoin de passer autant de temps en laboratoire pour les tests et la caractérisation. Comme la méthode peut en principe être appliquée à toutes les classes de protéines, nous pouvons désormais apprécier de manière plus ciblée les détails complexes du fonctionnement des protéines".
Note: Cet article a été traduit à l'aide d'un système informatique sans intervention humaine. LUMITOS propose ces traductions automatiques pour présenter un plus large éventail d'actualités. Comme cet article a été traduit avec traduction automatique, il est possible qu'il contienne des erreurs de vocabulaire, de syntaxe ou de grammaire. L'article original dans Anglais peut être trouvé ici.