Microscopie de fluorescence à résolution Ångström
Cette innovation est sur le point d'inaugurer un changement de paradigme dans notre approche de l'étude des systèmes biologiques avec un niveau de détail jusqu'à présent inégalé
Le groupe de recherche de Ralf Jungmann, de l'Institut Max Planck de biochimie (MPIB) et de la Ludwig-Maximilians-Universität (LMU) de Munich, a réalisé une percée dans le domaine de la microscopie à fluorescence. L'équipe a mis au point l'amélioration de la résolution par imagerie séquentielle (RESI), une technique révolutionnaire qui améliore la résolution de la microscopie à fluorescence jusqu'à l'échelle d'Ångström. Cette innovation est sur le point d'entraîner un changement de paradigme dans notre approche de l'étude des systèmes biologiques avec un niveau de détail jusqu'ici inégalé.
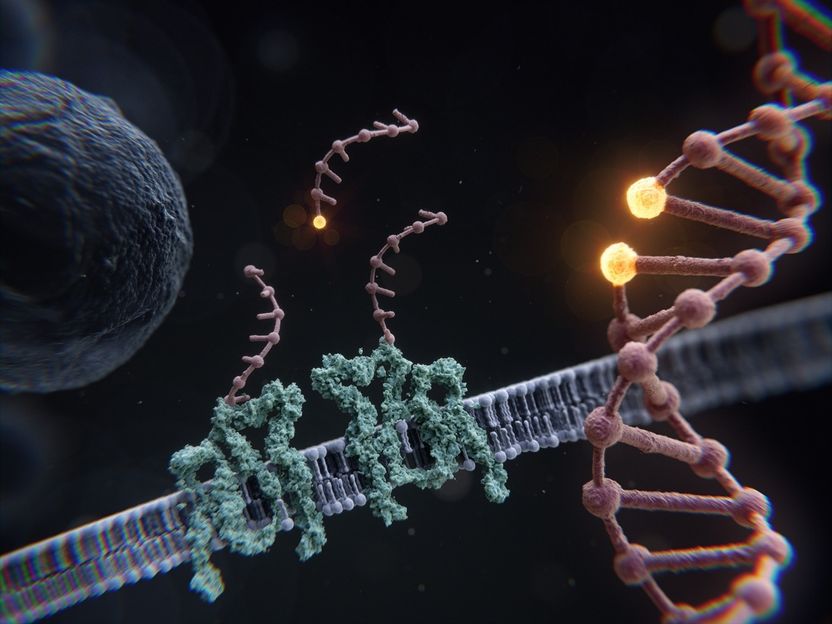
RESI permet la microscopie sur des échelles de longueur avec une résolution de l'ordre de l'Ångström : Des cellules entières aux protéines individuelles, en passant par la distance entre deux bases adjacentes de l'ADN.
Illustration: Max Iglesias, MPI für Biochemie
Les cellules, unités fondamentales de la vie, contiennent une pléthore de structures, de processus et de mécanismes complexes qui soutiennent et perpétuent les systèmes vivants. De nombreux composants cellulaires essentiels, tels que l'ADN, l'ARN, les protéines et les lipides, ne mesurent que quelques nanomètres. Ils sont donc beaucoup plus petits que la limite de résolution de la microscopie optique traditionnelle. La composition et l'agencement exacts de ces molécules et structures sont donc souvent inconnus, ce qui entraîne un manque de compréhension mécaniste des aspects fondamentaux de la biologie.
Ces dernières années, les techniques de super-résolution ont fait des progrès considérables pour résoudre de nombreuses structures subcellulaires en dessous de la limite de diffraction classique de la lumière. La microscopie de localisation à molécule unique, ou SMLM, est une approche de super-résolution qui permet de résoudre des structures de l'ordre de dix nanomètres en séparant temporellement leur émission de fluorescence individuelle. Comme les cibles individuelles s'illuminent stochastiquement (elles clignotent) dans un champ de vision par ailleurs sombre, leur emplacement peut être déterminé avec une précision inférieure à la diffraction. DNA-PAINT, inventé par le groupe Jungmann, est une technique SMLM qui utilise l'hybridation transitoire de brins d'ADN "imageur" marqués au colorant avec leurs compléments liés à la cible pour obtenir le clignotement nécessaire à la super-résolution. Toutefois, à ce jour, même DNA-PAINT n'a pas été en mesure de résoudre les plus petites structures cellulaires.
Dans l'étude actuelle menée par les coauteurs Susanne Reinhardt, Luciano Masullo, Isabelle Baudrexel et Philipp Steen en collaboration avec Jungmann, l'équipe présente une nouvelle approche de la microscopie à super-résolution qui permet une résolution spatiale fondamentalement "illimitée". La nouvelle technique, appelée "Resolution Enhancement by Sequential Imaging" (RESI), capitalise sur la capacité de DNA-PAINT à coder l'identité des cibles par le biais de séquences d'ADN uniques. En marquant des cibles adjacentes, trop proches les unes des autres pour être résolues même par la microscopie à super-résolution, avec des brins d'ADN différents, un degré supplémentaire de différenciation (un code-barres) est introduit dans l'échantillon. L'imagerie séquentielle d'une séquence, puis de l'autre (et donc de la cible) permet de les séparer sans ambiguïté. De plus, comme elles sont imagées séquentiellement, les cibles peuvent être arbitrairement proches l'une de l'autre, ce qu'aucune autre technique ne peut résoudre. En outre, RESI ne nécessite pas d'instrumentation spécialisée ; en fait, elle peut être appliquée à l'aide de n'importe quel microscope à fluorescence standard, ce qui la rend facilement accessible à la quasi-totalité des chercheurs.
Pour démontrer le bond en avant de RESI en matière de résolution, l'équipe s'est lancé le défi de résoudre l'une des plus petites distances spatiales dans un système biologique : La séparation entre les bases individuelles le long d'une double hélice d'ADN, espacées de moins d'un nanomètre (un milliardième de mètre). En concevant une nanostructure d'origami d'ADN qui présente des séquences d'ADN à brin unique dépassant d'une double hélice à une distance d'une paire de bases, puis en prenant des images séquentielles de ces brins uniques, l'équipe de recherche a résolu une distance de 0,85 nm (ou 8,5 Ångström) entre les bases adjacentes, un exploit inimaginable jusqu'à présent. Les chercheurs ont réalisé ces mesures avec une précision de 1 Ångström, soit un dix-milliardième de mètre, ce qui souligne les capacités sans précédent de l'approche RESI.
Il est important de noter que cette technique est universelle et ne se limite pas aux applications dans les nanostructures d'ADN. À cette fin, l'équipe a étudié le mode d'action moléculaire du Rituximab, un anticorps monoclonal anti-CD20 qui a été approuvé pour la première fois en 1997 pour le traitement du cancer du sang CD20-positif. Toutefois, l'étude des effets de ces molécules médicamenteuses sur les récepteurs moléculaires dépasse les capacités de résolution spatiale des techniques de microscopie traditionnelles. Comprendre si et comment ces schémas changent dans la santé et la maladie, ainsi qu'en cas de traitement, n'est pas seulement important pour la recherche mécanistique fondamentale, mais aussi pour la conception de nouvelles thérapies ciblées contre les maladies. Grâce à RESI, Jungmann et son équipe ont pu révéler l'arrangement naturel des récepteurs CD20 dans les cellules non traitées sous forme de dimères et découvrir comment le CD20 se réarrangeait en chaînes de dimères sous l'effet d'un traitement médicamenteux. Les résultats obtenus au niveau de la protéine unique permettent maintenant de faire la lumière sur le mode d'action moléculaire du Rituximab.
La RESI étant réalisée dans des cellules entières et intactes, elle comble le fossé entre les techniques purement structurales telles que la cristallographie aux rayons X ou la microscopie électronique cryogénique et les approches traditionnelles d'imagerie de cellules entières à plus faible résolution. Jungmann et son équipe sont convaincus que "cette technique sans précédent change véritablement la donne, non seulement pour la super-résolution, mais aussi pour la recherche biologique dans son ensemble".
Note: Cet article a été traduit à l'aide d'un système informatique sans intervention humaine. LUMITOS propose ces traductions automatiques pour présenter un plus large éventail d'actualités. Comme cet article a été traduit avec traduction automatique, il est possible qu'il contienne des erreurs de vocabulaire, de syntaxe ou de grammaire. L'article original dans Anglais peut être trouvé ici.