Une avancée majeure dans l'analyse des données ARN unicellulaires
Des chercheurs ont mis au point un outil informatique capable d'effectuer une analyse comparative précise d'ensembles complexes de données de séquençage unicellulaires.
Grâce aux progrès des études biologiques à haut débit, il est désormais possible de déterminer les gènes qui sont actifs dans une seule cellule. Cependant, l'analyse des ensembles de données complexes qui en résultent peut être difficile. Une équipe de l'université d'Osaka a mis au point CAPITAL, un nouvel outil informatique permettant de comparer des ensembles de données complexes provenant de cellules uniques.
Image symbolique
Computer generated picture
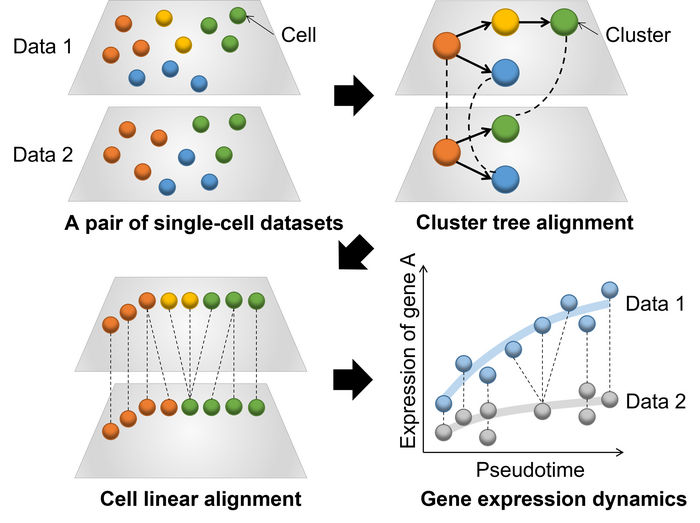
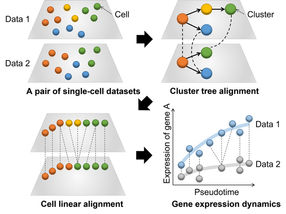
Vue d'ensemble de CAPITAL : un algorithme pour comparer les trajectoires pseudo-temporelles avec des branches
Yuki Kato
Le séquençage de l'ARN fournit des informations sur le sous-ensemble de la population entière de gènes qui sont activement exprimés, ou qui sont "activés". Grâce aux progrès technologiques, il est devenu possible de séquencer la population d'ARN d'une seule cellule. Cela peut fournir une grande quantité d'informations sur les changements spécifiques dans l'expression des gènes impliqués lorsqu'une grande population de cellules mixtes subit des processus dynamiques et transitoires, tels que la différenciation ou la mort cellulaire, parce que chaque cellule individuelle peut être analysée spécifiquement plutôt que de regrouper tous les différents types de cellules.
CAPITAL est spécifiquement conçu pour comparer des ensembles de données complexes provenant de cellules uniques subissant des processus de transition. Ces analyses sont effectuées en définissant une "trajectoire pseudo temporelle", qui place les cellules le long d'un chemin hypothétique qui reflète leur progression dans le processus de transition. Ces trajectoires ne sont pas toujours directes et linéaires ; elles peuvent devenir très complexes et ramifiées. Par le passé, seules les trajectoires linéaires pouvaient être alignées pour être comparées, mais l'innovation de l'équipe signifie que les trajectoires complexes et ramifiées peuvent désormais être alignées et comparées avec précision et automatiquement.
Après avoir développé l'algorithme utilisé pour CAPITAL, qui met en œuvre une méthode connue sous le nom d'alignement d'arbres, ils l'ont testé sur des ensembles de données synthétiques et des ensembles de données authentiques provenant de cellules de moelle osseuse. Les résultats ont démontré que CAPITAL est statistiquement plus précis et robuste que les algorithmes de calcul qui existaient auparavant, montrant des avancées majeures par rapport à ces méthodes.
La comparaison des trajectoires est une analyse puissante qui peut, par exemple, identifier la dynamique de l'expression des gènes entre différentes espèces pour fournir des informations sur les processus évolutifs. "Nous avons montré dans cette étude que CAPITAL peut révéler l'existence de modèles moléculaires différents entre les humains et les souris, même lorsque les modèles d'expression sont similaires et semblent être conservés", explique l'auteur principal Reiichi Sugihara. "Cela permettra d'identifier de nouveaux régulateurs qui déterminent le destin des cellules". Cette technologie n'est pas limitée à ce type de données, comme l'explique l'auteur principal Yuki Kato : "Notre nouvel outil de calcul peut être appliqué à un large éventail d'ensembles de données à haut débit, notamment des données pseudo-temporelles, spatiales et épigénétiques."
Cette nouvelle technique puissante permettra la comparaison globale des trajectoires de cellules uniques, ce qui pourrait conduire à l'identification de nouveaux gènes associés à des maladies qui n'ont pas pu être identifiés par des méthodes comparatives antérieures. Ainsi, CAPITAL représente une avancée significative dans le domaine de la biologie unicellulaire.
Note: Cet article a été traduit à l'aide d'un système informatique sans intervention humaine. LUMITOS propose ces traductions automatiques pour présenter un plus large éventail d'actualités. Comme cet article a été traduit avec traduction automatique, il est possible qu'il contienne des erreurs de vocabulaire, de syntaxe ou de grammaire. L'article original dans Anglais peut être trouvé ici.