Seguir y localizar a los miembros del microbioma vegetal con códigos de barras de ADN
Un equipo de investigación dirigido por Paul Schulze-Lefert, del Instituto Max Planck de Investigación en Fitomejoramiento de Colonia (Alemania), ha desarrollado un conjunto de herramientas modulares para rastrear las cepas bacterianas que colonizan el tejido vegetal en competencia con otros miembros del microbioma. El estudio se publica ahora en Nature Microbiology.
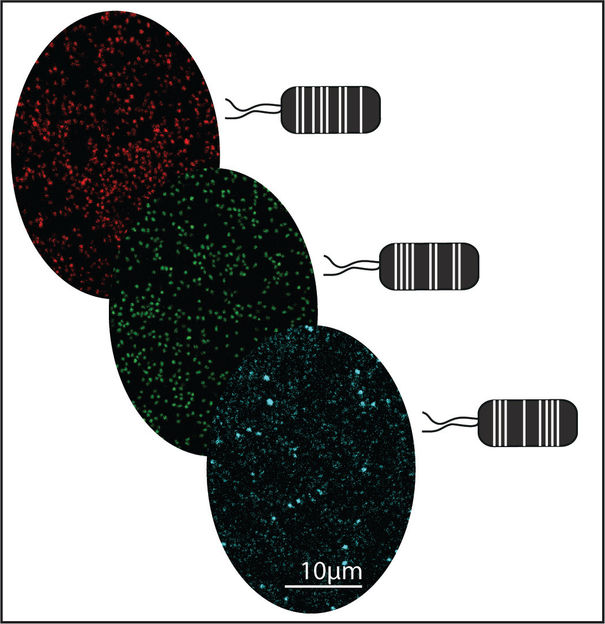
Esquema: Bacterias Pseudomonas capeferrum derivadas de plantas y marcadas con diferentes etiquetas fluorescentes utilizando el kit de herramientas MoBacTag.
Jana Ordon
La enorme diversidad de microorganismos que una planta sana alberga como comunidad en la naturaleza se denomina microbioma vegetal. Para evaluar la composición del microbioma, que es invisible a simple vista, se suele determinar la secuencia de ADN de un gen marcador microbiano universal formado por segmentos de secuencia variables y conservados. De este modo, las diferentes especies microbianas del microbioma pueden distinguirse entre sí en función de las secuencias polimórficas del marcador de ADN. Sin embargo, las actividades beneficiosas de los miembros del microbioma para la planta huésped, como la movilización de nutrientes minerales del suelo para su absorción por las raíces, a menudo sólo son llevadas a cabo por cepas microbianas individuales dentro de una especie bacteriana y dependen de la presencia de genes microbianos especializados. De ahí que los perfiles actuales de la microbiota basados en secuencias de ADN sean insuficientes para captar la verdadera diversidad genética de la comunidad microbiana del huésped.
Para superar esta limitación, los investigadores del Instituto Max Planck han desarrollado un conjunto de herramientas modulares que se utilizan como código de barras de ADN para las cepas bacterianas. Primero se inserta un código de barras de ADN en el cromosoma de una única cepa de un miembro del microbioma. En los análisis posteriores de los perfiles del microbioma en plantas, el código de barras de ADN se considera un gen marcador microbiano sintético. Además del código de barras de ADN cromosómico, también se incorporaron bloques de construcción genética para proteínas fluorescentes. Estas últimas permiten utilizar detectores de fluorescencia de alta sensibilidad para cartografiar dónde una cepa bacteriana con código de barras coloniza el tejido vegetal en competencia con otros miembros del microbioma.
A continuación, los investigadores llevaron a cabo experimentos con la bacteria promotora del crecimiento vegetal Pseudomonas capeferrum, que coloniza las raíces de la planta modelo Arabidopsis, así como con variantes de la bacteria que difieren de la cepa de tipo silvestre únicamente en la ausencia de determinados genes. Los genes de Pseudomonas correspondientes reducen la respuesta inmunitaria de la planta huésped y aumentan así la capacidad de la bacteria para colonizar las raíces de las plantas, lo que a su vez incrementa su actividad promotora del crecimiento vegetal. Las bacterias Pseudomonas marcadas con códigos de barras de ADN mostraron la capacidad diferencial esperada para colonizar las raíces de Arabidopsis cuando se inocularon plantas libres de gérmenes con cepas individuales. Lo sorprendente, sin embargo, fue la aparición de características cualitativamente nuevas de las bacterias Pseudomonas cuando se inocularon en la planta huésped combinaciones de la cepa de tipo salvaje y sus variantes junto con un consorcio microbioma de diferentes bacterias reunidas en el laboratorio. En biología, este fenómeno también se conoce como propiedad emergente o propiedad del sistema.
Por tanto, el uso de códigos de barras de ADN no sólo confirmó resultados anteriores, sino que también reveló nuevas actividades de los genes bacterianos que no podrían haberse identificado con métodos convencionales. El conjunto de herramientas modulares de códigos de barras de ADN puede utilizarse ahora en la investigación del microbioma para investigar la contribución de los genes microbianos individuales en el contexto de las comunidades microbianas, no sólo en las plantas, sino también en los estudios del microbioma de los animales.
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.