El método pionero revela la estructura dinámica del VIH
La técnica puede rastrear las moléculas en tiempo real, a temperatura ambiente
Los virus dan miedo. Invaden nuestras células como ejércitos invisibles, y cada tipo trae su propia estrategia de ataque. Mientras que los virus devastan comunidades de humanos y animales, los científicos luchan por defenderse. Muchos utilizan la microscopía electrónica, una herramienta que puede "ver" lo que las moléculas individuales del virus están haciendo. Sin embargo, incluso la tecnología más sofisticada requiere que la muestra sea congelada e inmovilizada para obtener la mayor resolución.
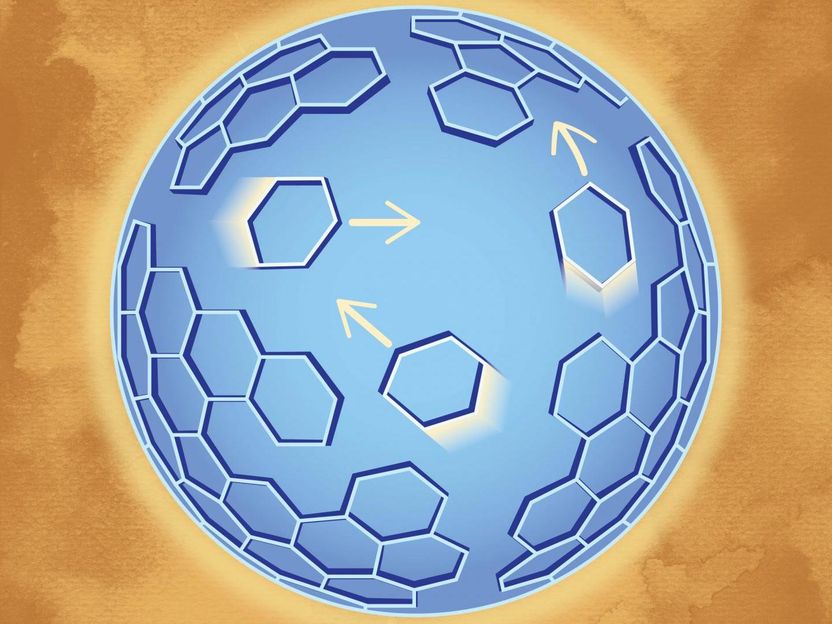
La interpretación de un artista de las proteínas de las moléculas Gag que forman una estructura hexagonal enrejada que se difunde a través de las partículas parecidas a un virus.
Dave Meikle/Saffarian Lab
Ahora, los físicos de la Universidad de Utah han sido pioneros en una forma de visualizar partículas similares a virus en tiempo real, a temperatura ambiente, con una resolución impresionante. En un nuevo estudio, el método revela que el entramado, que forma el principal componente estructural del virus de la inmunodeficiencia humana (VIH), es dinámico. El descubrimiento de un entramado difusor hecho de las proteínas Gag y GagPol, que durante mucho tiempo se consideró completamente estático, abre posibles nuevas terapias.
Cuando las partículas del VIH brotan de una célula infectada, los virus experimentan un retraso antes de ser infecciosos. La proteasa, una enzima que está incrustada como media molécula en las proteínas GagPol, debe unirse a otras moléculas similares en un proceso llamado dimerización. Esto desencadena la maduración viral que conduce a las partículas infecciosas. Nadie sabe cómo se encuentran y dimerizan estas moléculas de media proteasa, pero puede tener que ver con el reordenamiento de la red formada por las proteínas Gag y GagPol que se encuentran justo dentro de la envoltura viral. Gag es la principal proteína estructural y se ha demostrado que es suficiente para ensamblar partículas similares a las del virus. Las moléculas Gag forman una estructura hexagonal enrejada que se entrelaza consigo misma con minúsculos huecos intercalados. El nuevo método demostró que el entramado de la proteína Gag no es estático.
"Este método está un paso adelante al utilizar la microscopía que tradicionalmente sólo da información estática. Además de los nuevos métodos de microscopía, utilizamos un modelo matemático y experimentos bioquímicos para verificar la dinámica de la red", dijo la autora principal Ipsita Saha, asistente de investigación de posgrado en el Departamento de Física y Astronomía de la U. "Aparte del virus, una de las principales implicaciones del método es que puedes ver cómo se mueven las moléculas en una célula. Puedes estudiar cualquier estructura biomédica con esto."
Mapeo de una nanomáquina
Los científicos no buscaban estructuras dinámicas al principio... sólo querían estudiar el entramado de la proteína Gag. Saha dirigió el esfuerzo de dos años para "hackear" las técnicas de microscopía para poder estudiar las partículas del virus a temperatura ambiente para observar su comportamiento en la vida real. La escala del virus es minúscula, unos 120 nanómetros de diámetro, por lo que Saha utilizó la microscopía de localización fotoactivada interferométrica (iPALM).
Primero, Saha marcó la Gag con una proteína fluorescente llamada Dendra2 y produjo partículas parecidas a virus de las proteínas Gag-Dendra2 resultantes. Estas partículas parecidas al virus son las mismas que las partículas del VIH, pero hechas sólo de la estructura de la red de proteínas Gag-Dendra2. Saha mostró que las proteínas Gag-Dendra2 resultantes ensamblaron las partículas similares al virus de la misma manera que las partículas similares al virus formaron las proteínas Gag normales. El aditamento fluorescente permitió a iPALM visualizar la partícula con una resolución de 10 nanómetros. Los científicos descubrieron que cada partícula inmovilizada con apariencia de virus incorporaba entre 1400 y 2400 proteínas Gag-Dendra2 dispuestas en una red hexagonal. Cuando utilizaron los datos del iPALM para reconstruir una imagen en lapso de tiempo de la red, parecía que la red de Gag-Dendra2 no era estática en el tiempo. Para asegurarse, lo verificaron independientemente de dos maneras: matemáticamente y bioquímicamente.
Primero, dividieron la red de proteínas en segmentos uniformes y separados. Usando un análisis de correlación, probaron cómo cada segmento se correlacionaba consigo mismo a lo largo del tiempo, de 10 a 100 segundos. Si cada segmento continuaba correlacionándose con él mismo, las proteínas eran estacionarias. Si perdían la correlación, las proteínas se habían difundido. Descubrieron que con el tiempo, las proteínas eran bastante dinámicas.
La segunda forma en que verificaron la red dinámica fue bioquímicamente. Para este experimento, crearon partículas parecidas a virus cuyo entramado consistía en un 80% de proteínas de tipo salvaje Gag, un 10% de Gag etiquetadas con SNAP, y un 10% de Gag etiquetadas con Halo. SNAP y Halo son proteínas que pueden unirse a un enlazador que las une para siempre. La idea era identificar si las moléculas en la red de proteínas permanecían estacionarias, o si migraban de posición.
"Las proteínas Gag se ensamblan al azar. Las moléculas SNAP y Halo podrían estar en cualquier lugar dentro de la red, algunas pueden estar cerca una de la otra y otras estarán muy lejos", dijo Saha. "Si la red cambia, hay una posibilidad de que las moléculas se acerquen unas a otras".
Saha introdujo una molécula llamada Haxs8 en las partículas similares al virus. Haxs8 es un dímero, una molécula que une covalentemente las proteínas SNAP y Halo cuando están en un radio de unión entre ellas. Si las moléculas SNAP o Halo se mueven una al lado de la otra, producirán un complejo dimerizado. Rastreó estas concentraciones de complejos dimerizados a lo largo del tiempo. Si la concentración cambiara, indicaría que nuevos pares de moléculas se encuentran entre sí. Si la concentración disminuyera, indicaría que las proteínas se separaron. De cualquier manera, indicaría que se había producido un movimiento. Encontraron que con el tiempo, el porcentaje del complejo dimerizado aumentó; las proteínas HALO y SNAP Gag se movían por toda la red y se unían con el tiempo.
Una nueva herramienta para estudiar los virus
Este es el primer estudio que muestra que la estructura de la red de proteínas de un virus envuelto es dinámica. Esta nueva herramienta será importante para entender mejor los cambios que ocurren dentro de la red a medida que las nuevas partículas del virus pasan de inmaduras a peligrosamente infecciosas.
"¿Cuáles son los mecanismos moleculares que conducen a la infección? Abre una nueva línea de estudio", dijo Saha. "Si puedes averiguar ese proceso, tal vez puedas hacer algo para evitar que se encuentren, como un tipo de droga que detenga al virus en su camino".
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.






































