Un nuevo método para estudiar la función de los ribosomas
Científicos de la UIC utilizan la ligadura química nativa para fusionar péptidos con ARNt
En el interior de unas diminutas máquinas celulares llamadas ribosomas, las cadenas de material genético llamadas ARN mensajeros (ARNm) se emparejan con los correspondientes ARN de transferencia (ARNt) para crear secuencias de aminoácidos que salen del ribosoma en forma de proteínas. Las proteínas inacabadas se denominan cadenas nacientes y quedan unidas al ribosoma.
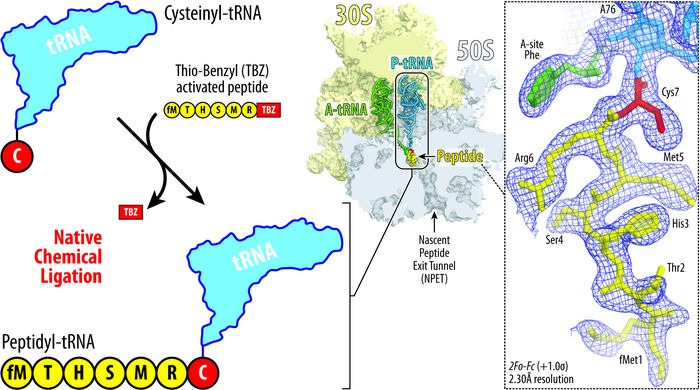
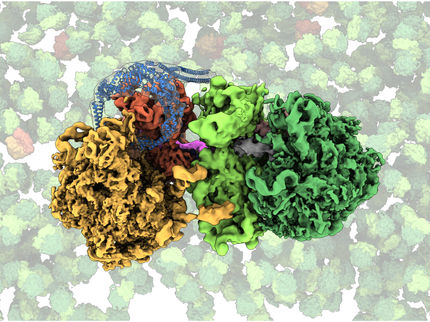
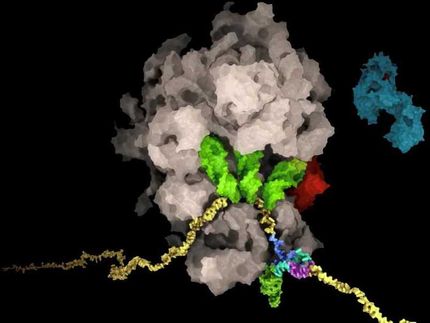
Ilustración que muestra el principio del enfoque de ligadura química nativa desarrollado por Syroegin, et al. La adición del aminoácido cisteína (rojo) al ARNt (azul, arriba a la izquierda) permite que el ARNt se fusione con un péptido (amarillo, abajo a la izquierda). La estructura del ribosoma resultante (centro) y los mapas de densidad electrónica capturados para el peptidil-ARNt dentro del ribosoma (derecha) se obtuvieron mediante cristalografía de rayos X en los experimentos UIC.
Syroegin, et al.
Los científicos saben que algunas de estas cadenas nacientes pueden regular la actividad del ribosoma y que, en ocasiones, las cadenas nacientes pueden interferir con los antibióticos, muchos de los cuales actúan dirigiéndose a la actividad del ribosoma bacteriano. Los científicos no saben por qué ocurre esto, principalmente porque es difícil visualizar cómo son las interacciones ribosoma-péptido-fármaco mientras las proteínas inacabadas están todavía unidas al ribosoma.
Ahora, los científicos de la Universidad de Illinois Chicago son los primeros en informar de un método para la fijación estable de péptidos a los ARNt, lo que les ha permitido obtener nuevos conocimientos fundamentales sobre la función del ribosoma al determinar las estructuras a nivel atómico de los ribosomas y las formas que adoptan estos péptidos dentro del ribosoma.
"El reto ha sido ver de cerca la estructura del ribosoma y el túnel de salida en presencia de los péptidos nacientes porque, en la naturaleza, el ribosoma es muy rápido para que podamos captar imágenes o realizar experimentos", dijo Yury Polikanov, profesor asociado del departamento de ciencias biológicas de la Facultad de Artes Liberales y Ciencias. "Hasta la llegada de este nuevo método, esencialmente hemos estado ciegos para ver lo que está sucediendo en el sitio activo del ribosoma en este momento crítico".
Polikanov y su colega Egor Syroegin, candidato a doctor en ciencias biológicas de la UIC, utilizaron un método denominado ligadura química nativa para fusionar péptidos personalizados con el ARNt y obtener lo que se denomina un peptidil-ARNt.
"La obtención de moléculas de ARNt unidas a péptidos, similares a las que se encuentran en el interior del ribosoma durante la síntesis de proteínas, ha sido el sueño de muchos investigadores en este campo durante casi dos décadas", dijo Polikanov. "Esto ha sido extremadamente difícil porque no hay enzimas que puedan unir directamente péptidos a un ARNt".
"El método se ha utilizado durante mucho tiempo en química, pero nunca se había aplicado de esta manera. Se trata de imitar a la naturaleza, básicamente, y con nuestra experiencia avanzada en la obtención de imágenes, ahora estamos viendo cómo funciona la naturaleza a una alta resolución", dijo Syroegin.
Con este nuevo enfoque, Polikanov y Syroegin determinaron un conjunto de estructuras de alta resolución del ribosoma que lleva peptidil-ARNt de varias longitudes.
El análisis detallado de estas estructuras proporciona nuevos y sorprendentes conocimientos sobre el mecanismo del centro catalítico del ribosoma y responde a varias cuestiones fundamentales que se plantean desde hace tiempo en el campo de los ribosomas, dijo Polikanov.
"Vimos que, dependiendo de la secuencia, diferentes péptidos pueden formar diferentes formas o pliegues dentro del túnel ribosómico, y podemos sintetizar diferentes péptidos de diferentes secuencias y luego seguir su forma con mucha precisión, debido a la alta resolución de nuestras estructuras", dijo Syroegin. "Así que ahora podemos decir con mucha seguridad que 'estos péptidos, de esta secuencia, tienen esta forma' u 'otro péptido tiene otra forma'. Eso es importante porque el plegamiento del péptido naciente determina si los fármacos detendrían el ribosoma o no".
"Este método abre innumerables vías para los estudios estructurales y funcionales destinados a comprender los mecanismos de funcionamiento del ribosoma, así como la detención del ribosoma por secuencias inducida por ciertos antibióticos", dijo Polikanov.
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.
Publicación original
"Insights into the ribosome function from the structures of non-arrested ribosome–nascent chain complexes"; Nature Chemistry.