Une nouvelle méthode pour étudier la fonction des ribosomes
Des scientifiques de l'UIC utilisent la ligature chimique native pour fusionner des peptides à des ARNt
À l'intérieur de minuscules machines cellulaires appelées ribosomes, des chaînes de matériel génétique appelées ARN messagers (ARNm) sont mises en correspondance avec les ARN de transfert (ARNt) correspondants pour créer des séquences d'acides aminés qui sortent du ribosome sous forme de protéines. Les protéines inachevées sont appelées chaînes naissantes et restent attachées au ribosome.
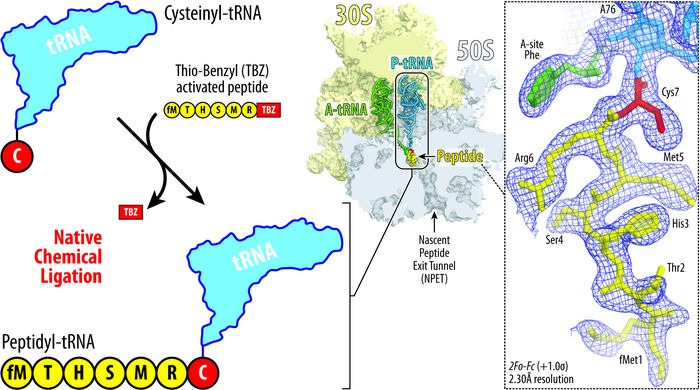
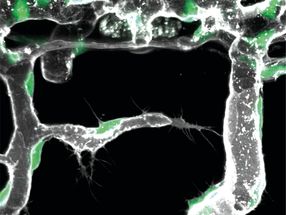
Illustration montrant le principe de l'approche de ligature chimique native développée par Syroegin, et al. L'ajout de l'acide aminé cystéine (rouge) à l'ARNt (bleu, en haut à gauche) permet à l'ARNt de fusionner avec un peptide (jaune, en bas à gauche). La structure du ribosome qui en résulte (au milieu) et les cartes de densité électronique capturées pour le peptidyl-tRNA à l'intérieur du ribosome (à droite) ont été obtenues par cristallographie aux rayons X dans les expériences de l'UIC.
Syroegin, et al.
Les scientifiques savent que certaines de ces chaînes naissantes peuvent réguler l'activité du ribosome et que les chaînes naissantes peuvent parfois interférer avec les antibiotiques - dont beaucoup agissent en ciblant l'activité du ribosome bactérien. Les scientifiques ne savent pas pourquoi cela se produit, principalement parce qu'il est difficile de visualiser les interactions ribosome-peptide-médicament lorsque les protéines inachevées sont encore attachées au ribosome.
Aujourd'hui, des scientifiques de l'université de l'Illinois à Chicago sont les premiers à avoir découvert une méthode de fixation stable des peptides aux ARNt, ce qui leur a permis d'acquérir de nouvelles connaissances fondamentales sur la fonction du ribosome en déterminant les structures des ribosomes au niveau atomique et les formes que prennent ces peptides à l'intérieur du ribosome.
"Le défi a été de voir de près la structure du ribosome et du tunnel de sortie en présence des peptides naissants car, dans la nature, le ribosome est très rapide pour que nous puissions capturer des images ou mener des expériences", a déclaré Yury Polikanov, professeur associé au département des sciences biologiques du College of Liberal Arts and Sciences. "Jusqu'à l'avènement de cette nouvelle méthode, nous avons essentiellement été aveuglés de voir ce qui se passe dans le site actif du ribosome à ce moment critique."
Polikanov et son collègue Egor Syroegin, candidat au doctorat en sciences biologiques à l'UIC, ont utilisé une méthode appelée ligature chimique native pour fusionner des peptides personnalisés avec l'ARNt afin d'obtenir ce qu'on appelle un ARNt peptidyle.
"Obtenir des molécules d'ARNt liées à des peptides, semblables à celles qui se trouvent à l'intérieur du ribosome lors de la synthèse des protéines, est resté le rêve de nombreux chercheurs dans ce domaine pendant près de deux décennies", a déclaré M. Polikanov. "Cela a été extrêmement difficile car il n'existe pas d'enzymes capables de fixer directement les peptides à un ARNt".
"La méthode est utilisée depuis longtemps en chimie, mais elle n'a jamais été appliquée de cette manière. Il s'agit d'imiter la nature, en gros, et grâce à notre expérience en imagerie avancée, nous voyons maintenant comment la nature fonctionne à haute résolution", a déclaré M. Syroegin.
Grâce à cette nouvelle approche, Polikanov et Syroegin ont déterminé un ensemble de structures haute résolution du ribosome portant des peptidyl-tRNA de différentes longueurs.
L'analyse détaillée de ces structures fournit des informations nouvelles et surprenantes sur le mécanisme du centre catalytique du ribosome et répond à plusieurs questions fondamentales de longue date dans le domaine des ribosomes, a déclaré Polikanov.
"Nous avons vu que, selon la séquence, différents peptides peuvent former différentes formes ou plis à l'intérieur du tunnel ribosomal, et nous pouvons synthétiser différents peptides de différentes séquences et ensuite suivre leur forme très précisément, en raison de la haute résolution de nos structures", a déclaré Syroegin. "Nous pouvons donc maintenant dire en toute confiance que 'ces peptides, de cette séquence, ont cette forme' ou 'un autre peptide a une autre forme'. C'est important car le repliement des peptides naissants détermine si les médicaments arrêteront ou non le ribosome."
"Cette méthode ouvre d'innombrables voies pour des études structurelles et fonctionnelles visant à comprendre les mécanismes de fonctionnement du ribosome, ainsi que le blocage du ribosome spécifique à une séquence induit par certains antibiotiques", a déclaré Polikanov.
Note: Cet article a été traduit à l'aide d'un système informatique sans intervention humaine. LUMITOS propose ces traductions automatiques pour présenter un plus large éventail d'actualités. Comme cet article a été traduit avec traduction automatique, il est possible qu'il contienne des erreurs de vocabulaire, de syntaxe ou de grammaire. L'article original dans Anglais peut être trouvé ici.
Publication originale
"Insights into the ribosome function from the structures of non-arrested ribosome–nascent chain complexes"; Nature Chemistry.