Predecir el comportamiento de una proteína desde su apariencia
Las proteínas son los componentes básicos de la vida y desempeñan un papel clave en todos los procesos biológicos. Comprender cómo interactúan con su entorno es, por lo tanto, vital para desarrollar una terapéutica eficaz y la base para el diseño de células artificiales.
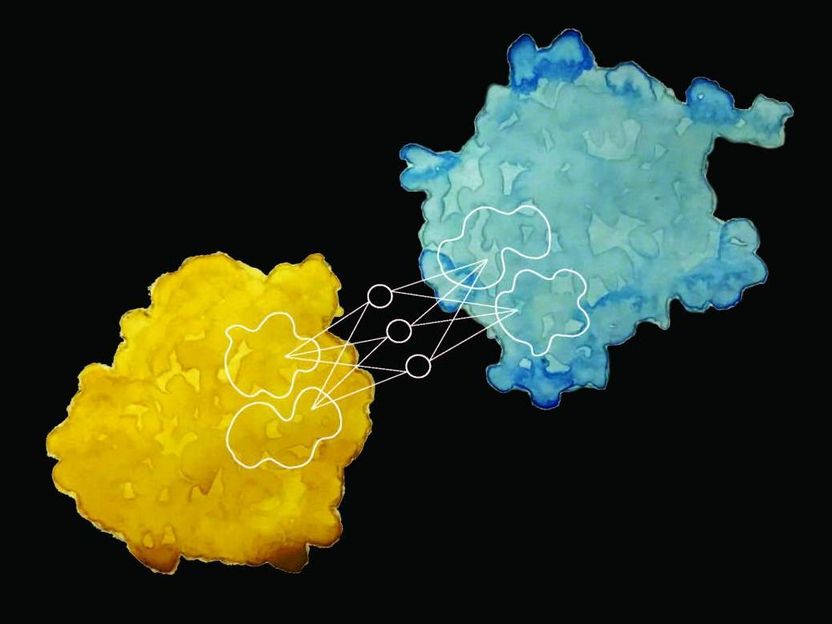
Los investigadores de la EPFL han desarrollado una nueva forma de predecir las interacciones de una proteína con otras proteínas y biomoléculas, y su actividad bioquímica, simplemente observando su superficie.
Laura Persat / 2019 EPFL
Investigadores del Laboratorio de Diseño de Proteínas e Inmunoingeniería (LPDI), parte del Instituto de Bioingeniería de la EPFL en la Escuela de Ingeniería, trabajando con colaboradores de la USI-Lugano, el Imperial College y la división de Investigación en Aprendizaje Gráfico de Twitter, han desarrollado una técnica innovadora de aprendizaje de máquinas para predecir estas interacciones y describir la actividad bioquímica de una proteína basada únicamente en la apariencia de la superficie. Además de profundizar nuestra comprensión de cómo funcionan las proteínas, el método, conocido como MaSIF, también podría apoyar el desarrollo de componentes basados en proteínas para las células artificiales del mañana.
Investigación basada en datos
Los investigadores tomaron un vasto conjunto de datos de la superficie de la proteína y alimentaron las propiedades químicas y geométricas en un algoritmo de aprendizaje por máquina, entrenándolo para que coincidiera con estas propiedades con patrones de comportamiento y actividad bioquímica particulares. Luego utilizaron los datos restantes para probar el algoritmo. "Al escanear la superficie de una proteína, nuestro método puede definir una huella dactilar, que luego puede ser comparada entre proteínas", dice Pablo Gainza, el primer autor del estudio.
El equipo encontró que las proteínas que realizan interacciones similares comparten "huellas digitales" comunes.
"El algoritmo puede analizar miles de millones de superficies de proteínas por segundo", dice el director de LPDI, Bruno Correia. "Nuestra investigación tiene implicaciones significativas para el diseño de proteínas artificiales, permitiéndonos programar una proteína para que se comporte de cierta manera simplemente alterando sus propiedades químicas y geométricas de la superficie".
El método, publicado en formato de código abierto, también podría utilizarse para analizar la estructura superficial de otros tipos de moléculas.
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.
Publicación original
P. Gainza, F. Sverrisson, F. Monti, E. Rodolà, D. Boscaini, M. M. Bronstein, and B. E. Correia; "Deciphering interaction fingerprints from protein molecular surfaces using geometric deep learning"; Nature Methods; 2019.
Noticias más leídas
Publicación original
P. Gainza, F. Sverrisson, F. Monti, E. Rodolà, D. Boscaini, M. M. Bronstein, and B. E. Correia; "Deciphering interaction fingerprints from protein molecular surfaces using geometric deep learning"; Nature Methods; 2019.
Organizaciones
Más noticias del departamento ciencias

Reciba la industria de las ciencias biológicas en su bandeja de entrada
No se pierda nada a partir de ahora: Nuestro boletín electrónico de biotecnología, productos farmacéuticos y ciencias de la vida le pone al día todos los martes y jueves. Las últimas noticias del sector, los productos más destacados y las innovaciones, de forma compacta y fácil de entender en su bandeja de entrada. Investigado por nosotros para que usted no tenga que hacerlo.