Diagnostik-Kits:
SepsiTest™-UMD CE IVD
Kultur-unabhängiger Nachweis von Bakterien und Pilzen in Blut/Körperflüssigkeiten und Gewebe
Anreicherung mikrobieller DNA und hoch sensitive 16S/18S PCR zur Erregeridentifizierung
SepsiTest™-UMD CE IVD ermöglicht einen schnellen Nachweis von Bakterien und Pilzen, unabhängig davon ob die Erreger wachsen können oder durch eine Antibiotikatherapie gehemmt sind beziehungsweise generell in den Medien nicht wachsen.
Durch den Abbau humaner und frei-zirkulierender mikrobieller DNA wird eine hohe Spezifität für den Nachweis bakterieller und fungaler DNA sichergestellt, und zwar nur in intakten Zellen. Der Nachweis erfolgt durch eine 16S beziehungsweise 18S rRNA-Gen PCR oder Real-Time PCR. Eine Sequenzierung mit BLAST-Analyse identifiziert den Erreger. Die Analyse ist somit eubakteriell beziehungsweise pan-fungal.
Applikation
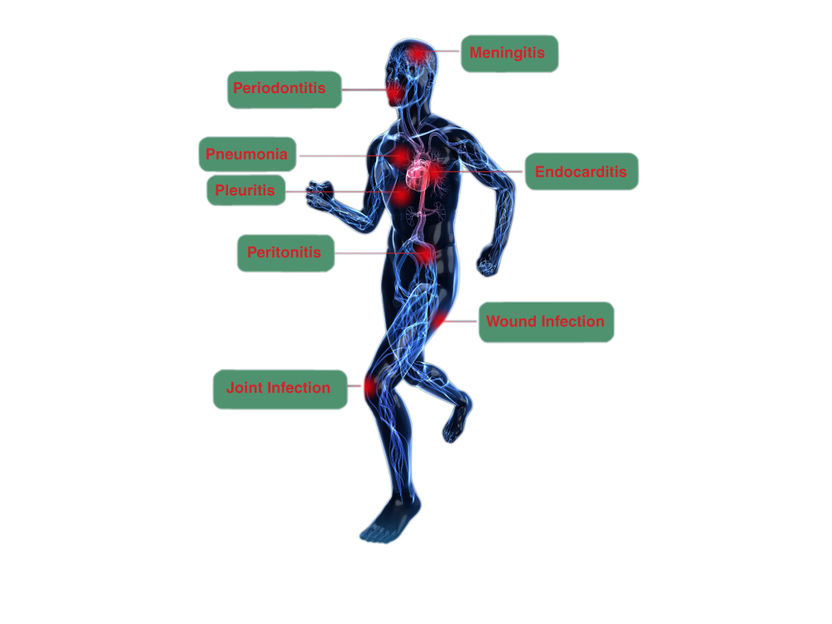
SepsiTest-UMD CE IVD ist besonders geeignet für Proben, die häufig in der Kultur negativ sind als Wirkung einer Antibiotikatherapie, z. B. bei Erkrankungen wie:
|
|
|
|
|
|
|
|
Handhabung/Laborausstattung
Für die Durchführung des SepsiTest-UMD CE IVD ist lediglich Standardausrüstung eines molekularbiologischen Labors erforderlich.
SepsiTest-UMD CE IVD beinhaltet alle für die DNA-Extraktion und PCR/Real-Time PCR benötigten Reagenzien und Verbrauchsmaterialien in besonders reiner und DNA-freier Qualität, sowie Primer für die Sanger-Sequenzierung.
Der Test umfasst folgende Schritte:
- Entfernung humaner DNA und freier DNA aus toten Bakterien und Pilzen
- Extraktion der DNA aus intakten Erregern
- 16S und 18S rRNA-Gen PCR oder -Real-Time PCR-Analyse
- Sequenzierung von Amplifikaten mit BLAST-Sequenzanalyse zur Identifizierung der Erreger.
Probenmaterial
Verschiedene klinische Probenmaterialien können mit dem SepsiTest-UMD CE IVD bearbeitet werden:
- Körperflüssigkeiten (Blut, CSF, Synovialflüssigkeit, Aszites, Peritonealpunktat, BAL etc.)
- Gewebe (Herzklappen, Knochen, Gehirn, andere Biopsien)
- Abstriche (Wunden, Gewebe, Mund, Nase etc.).
Neu: Add-On 10 CE IVD für die Analyse von bis zu 10 ml mit dem SepsiTest-UMD CE IVD.
Sensitivitäten
Die auf Spiking-Experimenten mit 1 ml-Blutproben basierte analytische Sensitivität wurde an verschiedenen Erregern und Stämmen getestet. Sie liegt beispielsweise für S. aureus bei 20 cfu/ml, für E. coli bei 40 cfu/ml und für C. albicans bei 10 cfu/ml.
SepsiTest-UMD CE IVD wurde in klinischen Studien evaluiert.
Automation
Zur Reduzierung von Hands-on Time und Arbeitskosten sowie zur Sicherung der Qualität der Probenvorbereitung stehen Lösungen mit teilautomatisierter (UMD-SelectNA™ CE-IVD) und vollautomatisierter DNA-Extraktion (Micro-Dx™) zur Verfügung.

1
SepsiTest-UMD CE IVD – Diagnose von Infektionserkrankungen

2
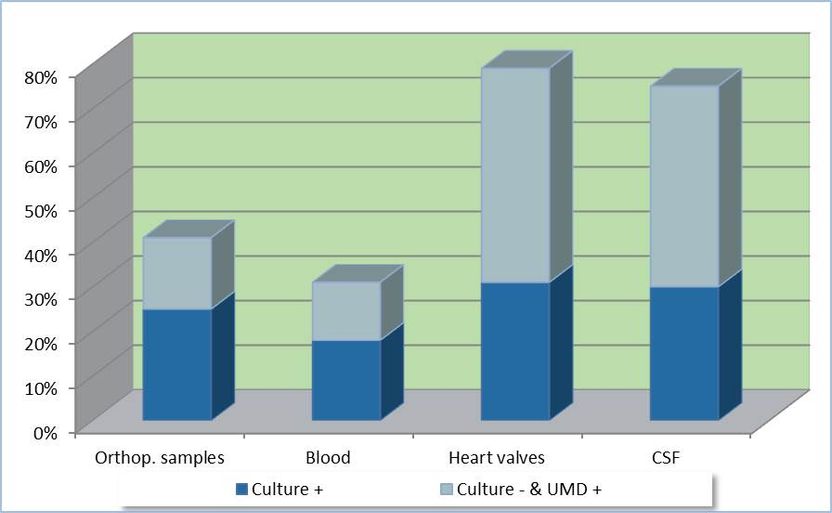
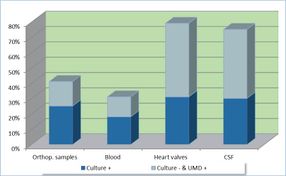
SepsiTest-UMD CE IVD – erhöht die Nachweisrate bei Kultur-negativen Infektionen

3
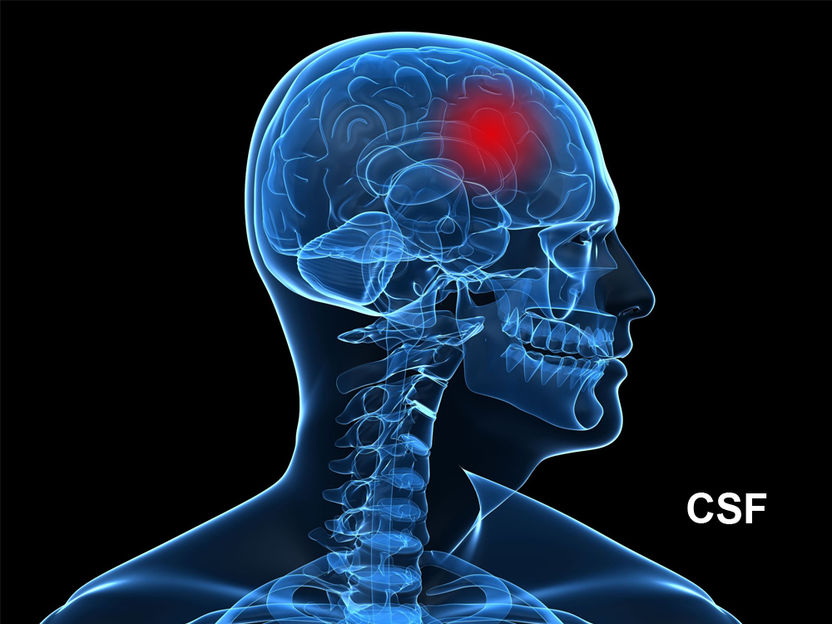
SepsiTest-UMD CE IVD – Diagnose von Bakterien und Pilzen in neurologischem Probenmaterial

4
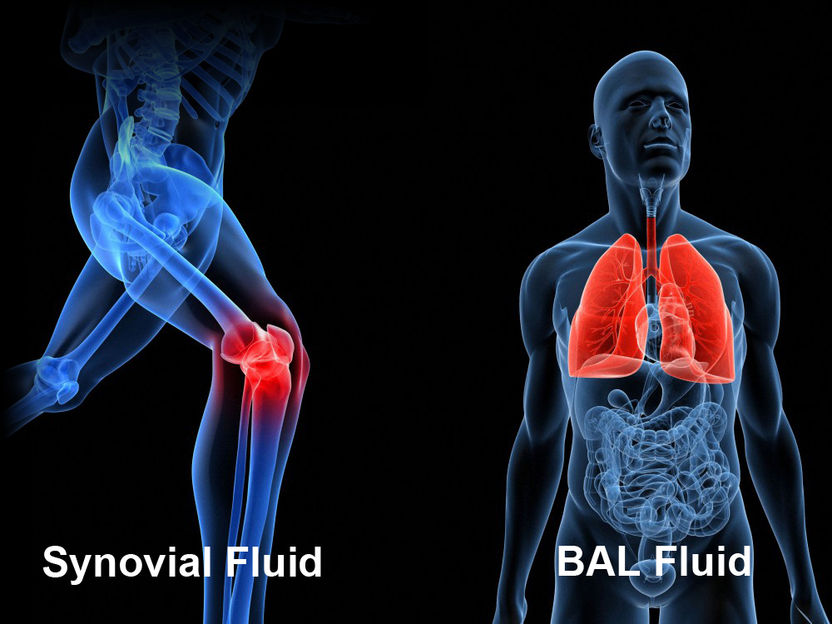
SepsiTest-UMD CE IVD – Diagnose von Bakterien und Pilzen bei orthopädischen und pulmonalen Infektionen

5
Universal Microbe Detection (UMD) – Diagnose von Bakterien und Pilzen bei orthopädischen und pulmonalen Infektionen
Produkteinordnung SepsiTest™-UMD CE IVD
Anwendungen
Produktkategorien
Jetzt Informationen zu SepsiTest™-UMD CE IVD anfordern

Diagnostik-Kits: SepsiTest™-UMD CE IVD
Kultur-unabhängiger Nachweis von Bakterien und Pilzen in Blut/Körperflüssigkeiten und Gewebe