Mikrobiologen identifizieren neue Proteinstruktur
Ein Forschungsteam aus Marburg und Frankfurt am Main hat ein neues Strukturelement identifiziert, das bei Enzymen vorkommt, mit denen Kohlendioxid in Biomoleküle eingebaut wird. Das neu entdeckte Muster kannte man bisher nur von künstlich hergestellten Molekülen, aber nicht von natürlich vorkommenden Proteinen.
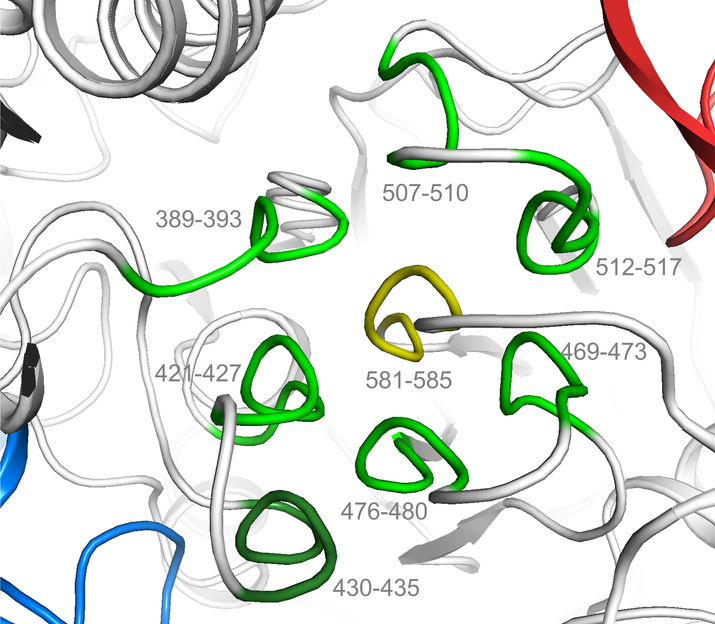
Das Bändermodell zeigt das neue Strukturmotiv in schematischer Darstellung: Eine zentrale Helix (gelb) ist dabei von sechs peripheren Helices umgeben (grün), während die achte Helix (dunkelgrün) das Muster nach außen fortsetzt.
Dr. Ulrich Ermler
Proteine bestehen aus Aminosäuren, die zu Ketten aneinander gereiht sind. Damit die Proteine ihre jeweilige Funktion erfüllen, ist es nicht nur von Belang, welche Aminosäuren sie enthalten und wie diese angeordnet sind – ebenso wichtig ist auch die dreidimensionale Form, in die sich die Kette faltet, die so genannte Sekundärstruktur. Dabei treten regelmäßige, wiederkehrende Strukturmotive auf. „Wir haben ein für Proteine neuartiges Strukturmotiv identifiziert“, erklärt der Marburger Mikrobiologe Professor Dr. Johann Heider, Mitverfasser der aktuellen Studie.
Das Team um Heider und seinen Frankfurter Kollegen Dr. Ulrich Ermler untersuchte eine Carboxylase – dabei handelt es sich um Enzyme, die Kohlendioxid in Biomoleküle einbauen, wie es etwa bei der Fotosynthese geschieht, aber auch bei mikrobiellen Abbauprozessen. Carboxylasen sind mithin Moleküle, die an den wichtigsten und am weitesten verbreiteten Vorgängen in der belebten Natur beteiligt sind.
Die Wissenschaftler nahmen sich die Acetophenon-Carboxylase vor, „einen besonders komplexen Vertreter der Enzymfamilie“, wie Heider betont. Die Gruppe entdeckte dabei Konstruktionsdetails, die bislang noch nicht als ein eigenständiges Strukturmotiv erkannt worden waren. Das Motiv besteht aus acht kurzen, schraubenartigen Formen, sogenannten Helices. Diese sind zu einem Bündel angeordnet, wobei eine zentrale Helix von sechs anderen umringt wird, während die achte das Muster nach außen fortsetzt.
„Dieses Motiv war bisher nur in künstlich hergestellten Glycin-reichen Polypeptiden bekannt, aber nicht in natürlich vorkommenden Proteinen“, hebt Heider hervor. Die detaillierte Beschreibung der dreidimensionalen Struktur gestattet es nun, näher zu untersuchen, wie die Acetophenon-Carboxylase ihre Funktion bei mikrobiellen Abbauprozessen erfüllt.
Originalveröffentlichung
Weitere News aus dem Ressort Wissenschaft

Holen Sie sich die Life-Science-Branche in Ihren Posteingang
Ab sofort nichts mehr verpassen: Unser Newsletter für Biotechnologie, Pharma und Life Sciences bringt Sie jeden Dienstag und Donnerstag auf den neuesten Stand. Aktuelle Branchen-News, Produkt-Highlights und Innovationen - kompakt und verständlich in Ihrem Posteingang. Von uns recherchiert, damit Sie es nicht tun müssen.